逆温度1の事後分布のサンプルからWBICを計算する
この記事は以下のツイートを拝見してやってみようと思いました。
#統計 #Baysian もしも「元論文の式(20)をβ₁=1, β₂=1/log nの場合に適用した公式を使ってWBICを計算すると事後分布のサンプルの違いによる分散が大きくなる」とか「直接逆温度1/log nの事後分布のサンプルを生成して計算した方が精度が高い」とかの情報を知っている人がいたら教えて下さい。
— 黒木玄 Gen Kuroki (@genkuroki) 2017年11月10日
ツイートで言及されている渡辺先生の論文は以下です。
- S Watanabe (2013) "A widely applicable Bayesian information criterion" Journal of Machine Learning Research 14 (Mar), 867-897 (pdf file)
この記事では、以前WAICとLOOCVの比較をした時に使った3つのモデル(重回帰、ロジスティック回帰、非線形回帰)において、「定義通りに算出したオリジナルのWBIC」と「近似式(上記論文の(20)式)で求めたWBIC」を比較してみました。
手法
case 1 重回帰
真のモデルは以下です。
あてはめたモデルは以下です。
データ点の数Nについては20,100を試しました。例としてN = 20の場合を説明します。まず乱数でデータX(すなわち)を生成します。次にその
Xの値を使ってYを生成しますが、以下の二つの場合について計算しました。
- 1) MCMCサンプルの出方の違いによる影響:
Yを1つだけ生成して固定し、MCMCの乱数の種を変えて200回推定を行い、・
のそれぞれの分布を確認した
- 2) データの出方の違いによる影響:
Yを1000通り生成し、および相対的な差である
の分布を確認した
事後分布の推定はStanで行いました。iter=11000, warmup=1000, chains=4で実行して合計40000個のMCMCサンプルを得ています。
case 2 ロジスティック回帰
手順は重回帰の場合と同じです。使用したモデルだけが異なります。真のモデルは以下です。
あてはめたモデルは以下です。
case 3 非線形回帰 ミカエリス・メンテン型
手順は重回帰の場合と同じです。使用したモデルだけが異なります。真のモデルは以下です。
あてはめたモデルは以下です。
case 3b 真のモデルが含まれない場合
あてはめたモデルが以下の場合も試しました。
結果
計算速度
Stanを使う場合、近似式のモデルの方がサンプリングが速いので、計算速度は近似式の方が少し速いです。どれだけ速くなるかはモデル依存で場合によりけりです。
MCMCサンプルの出方の違いによる影響
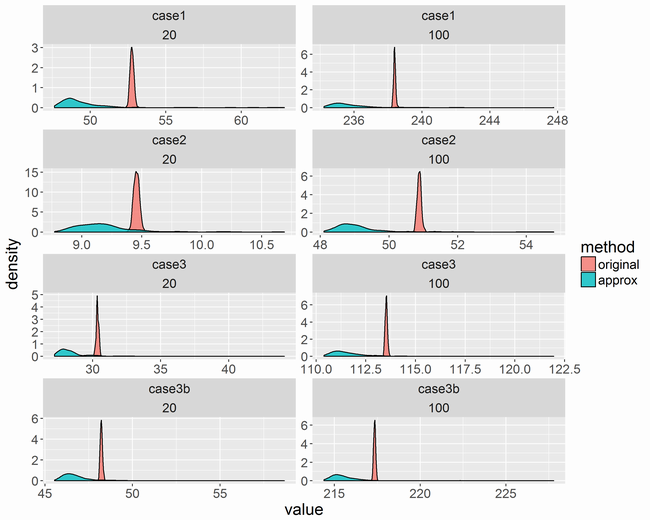
近似式の方はMCMCサンプルの出方によってかなりばらつくようです。また、少し値が低くなっています。
データの出方の違いによる影響
差
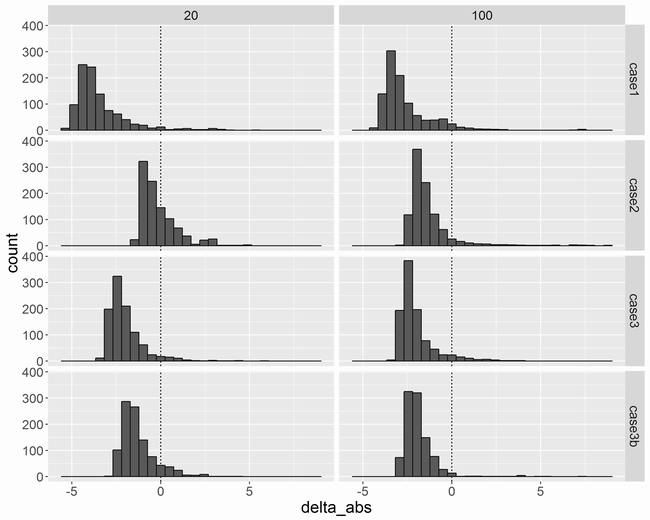
横軸はです。少しマイナスに偏った分布になりました。これが近似で捨てた項の影響なのか、Stanによるサンプリングの影響なのかは分かりません。
相対的な差
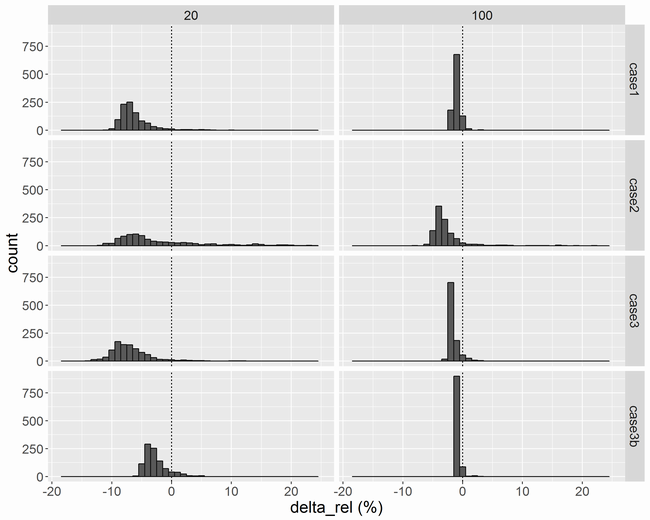
横軸は相対的な差であるです。ロジスティック回帰の
N = 20の場合は、しばしばが0に近くなるので尾を引いています。
Nが増えるに従って相対的な差は小さくなり、N = 100では±5%ぐらいに収まりそうです。
まとめ
Nが大きいときは近似式でスピードを重視しても大丈夫そう。でもNが小さいときは定義通り計算した方が無難に思えます。
ソースコード
case 1のソースコードを以下に載せます。
オリジナルのWBICを算出するためのStanコード
model/model1-ori.stanというファイル名とします。
data { int D; int N; matrix[N,D] X; vector[N] Y; } parameters { vector[D] b; real<lower=0> sigma; } transformed parameters { vector[N] mu; mu = X*b; } model { target += 1/log(N) * normal_lpdf(Y | mu, sigma); } generated quantities { vector[N] log_lik; for (n in 1:N) log_lik[n] = normal_lpdf(Y[n] | mu[n], sigma); }
近似式でWBICを算出するためのStanコード
model/model1-apx.stanというファイル名とします。
data { int D; int N; matrix[N,D] X; vector[N] Y; } parameters { vector[D] b; real<lower=0> sigma; } transformed parameters { vector[N] mu; mu = X*b; } model { Y ~ normal(mu, sigma); } generated quantities { vector[N] log_lik; for (n in 1:N) log_lik[n] = normal_lpdf(Y[n] | mu[n], sigma); }
各WBICを算出するRコード
library(rstan) wbic_original <- function(log_lik) { wbic <- - mean(rowSums(log_lik)) return(wbic) } wbic_approx <- function(log_lik) { b2 <- 1.0/log(ncol(log_lik)) b1 <- 1.0 log_denominator <- statnet.common::log_sum_exp(-(b2-b1)*(rowSums(-log_lik))) log_numerator <- statnet.common::log_sum_exp(-(b2-b1)*(rowSums(-log_lik)) + log(rowSums(-log_lik))) wbic <- exp(log_numerator - log_denominator) return(wbic) } set.seed(123) D <- 3 b <- c(1.3, -3.1, 0.7) SD <- 2.5 N <- 100 X <- cbind(1, matrix(runif(N*(D-1), -3, 3), N, (D-1))) Mu <- X %*% b Y <- rnorm(N, Mu, SD) data <- list(N=N, D=D, X=X, Y=Y) sm_ori <- stan_model(file='model/model1-ori.stan') sm_apx <- stan_model(file='model/model1-apx.stan') fit_ori <- sampling(sm_ori, pars='log_lik', data=data, iter=11000, warmup=1000, seed=123) fit_apx <- sampling(sm_apx, pars='log_lik', data=data, iter=11000, warmup=1000, seed=123) wbic_ori <- wbic_original(rstan::extract(fit_ori)$log_lik) wbic_apx <- wbic_approx(rstan::extract(fit_apx)$log_lik) c(wbic_ori=wbic_ori, wbic_apx=wbic_apx)
- 3~6行目:
を算出します。この記事参照。
- 8~15行目:
を算出します。途中で
{statnet.common}パッケージのlog_sum_exp関数を使っています。前の記事のようにStanに含まれるlog_sum_exp関数を使っても構いません(全く同じ数値になります)。 - 9行目:
log_likはN_mcmc×N(データの数)のmatrix型ですのでncol(log_lik)でNを取得しています。
渡辺先生の論文の(20)式の通りに計算しようとすると、expの内側が50ぐらい以上の数値になるため、計算が不安定になります。そのため(20)式の両辺の対数をとって計算して、最後にexpをかませて戻します。MCMCを使っている場合、(20)式の左辺の対数は以下のように式変形できます。
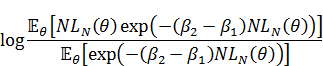
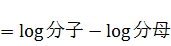
なので、まず易しい分母の方から計算すると、
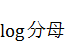
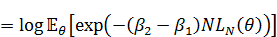
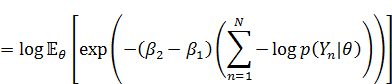
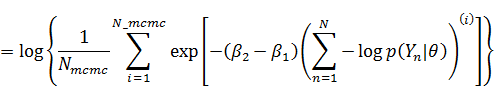
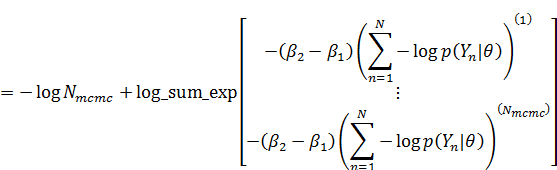
分子の方も同様に、
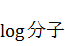
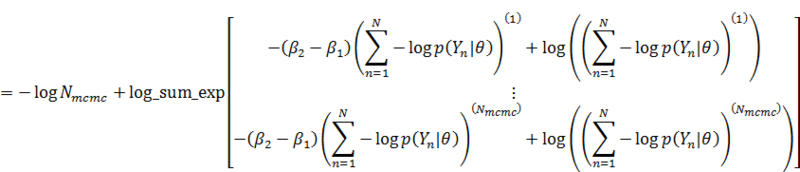
これをそのまま実装しています。
Enjoy!