ã“ã‚“ã«ã¡ã¯ã€‚サーãƒãƒ¼ã‚¨ãƒ³ã‚¸ãƒ‹ã‚¢ã®ä½è—¤å¤ªä¸€(@teach_kaiju)ã§ã™ã€‚
本記事ã§ã¯ç¤¾å†…ã§é–‹ç™ºã—ãŸã€æ•°å„„ã®ãƒ‡ãƒ¼ã‚¿ã‚’処ç†ã™ã‚‹ä»•çµ„ã¿ã‚’æä¾›ã™ã‚‹ gem MedPipe を紹介ã—ã¾ã™ã€‚
MedPipe ã¨ã¯
「Log ã®ãƒ‡ãƒ¼ã‚¿ã‚’å…¨ã¦å–å¾—ã—ã€ãƒ•ã‚©ãƒ¼ãƒžãƒƒãƒˆã—㦠tsv ã¨ã—㦠S3 ã«ã‚¢ãƒƒãƒ—ãƒãƒ¼ãƒ‰ã™ã‚‹ã€ã¨ã„ã†è¦ä»¶ãŒã‚ã£ãŸã¨ã—ã¾ã™ã€‚
ã“ã®è¦ä»¶ã‚’実ç¾ã™ã‚‹ãŸã‚ã«ã€ä¾‹ãˆã°ä»¥ä¸‹ã®ã‚ˆã†ãªå®Ÿè£…を考ãˆã‚‹ã“ã¨ãŒã§ãã¾ã™ã€‚
upload_file_name = "hoge_logs.csv" # 1. S3ã«ã‚¢ãƒƒãƒ—ãƒãƒ¼ãƒ‰ã™ã‚‹ãŸã‚ã® file ã‚’ç”¨æ„ Tempfile.create do |file| # 2. Log ã®ãƒ‡ãƒ¼ã‚¿ã‚’ DB ã‹ã‚‰å–å¾— HogeLog.find_each do |log| # 3. ãƒ•ã‚©ãƒ¼ãƒžãƒƒãƒˆå‡¦ç† formatted_data = format(log) # 4. ファイルã«æ›¸ã込㿠line = CSV.generate_line(formatted_data, col_sep: "\t") file.puts(line) end # 5. S3ã«ã‚¢ãƒƒãƒ—ãƒãƒ¼ãƒ‰ upload_s3(file, upload_file_name) end def format(log) # å‡¦ç† end def upload_s3(file, upload_file_name) # å‡¦ç† end
ãã‚Œã«å¯¾ã—ã¦ã€MedPipe を使ã†ã¨ä»¥ä¸‹ã®ã‚ˆã†ã«è¨˜è¿°ã§ãã¾ã™ã€‚
upload_file_name = "hoge_logs.csv" pipeline = MedPipe::Pipeline.new pipeline.apply(PipelineTask::HogeLogReader.new) # 1. Log ã®ãƒ‡ãƒ¼ã‚¿ã‚’ DB ã‹ã‚‰å–å¾— .apply(PipelineTask::HogeLogFormatter.new) # 2. ãƒ•ã‚©ãƒ¼ãƒžãƒƒãƒˆå‡¦ç† .apply(MedPipe::PipelineTask::TsvGenerater.new) # 3. ファイルã«æ›¸ã込㿠.apply(PipelineTask::S3Uploader.new(upload_file_name)) # 4. S3ã«ã‚¢ãƒƒãƒ—ãƒãƒ¼ãƒ‰ pipeline.run
ã“ã®ã‚ˆã†ã«ã€MedPipe を使ã†ã“ã¨ã§å‡¦ç†ã®æµã‚ŒãŒæ˜Žç¢ºã«ãªã‚Šã€å¯èªæ€§ã‚’å‘上ã•ã›ã‚‹ã“ã¨ãŒã§ãã¾ã™ã€‚
ãã‚Œã«åŠ ãˆã¦ä»¥ä¸‹ã®ã‚ˆã†ãªæ©Ÿèƒ½ã‚’容易ã«å®Ÿè£…ã™ã‚‹ã“ã¨ãŒã§ãã¾ã™ã€‚
- 並列処ç†
- クエリ最é©åŒ–ã®ãŸã‚ã®ã€in_batches を用ã„ãªã„独自データå–得処ç†
- 件数ã®ã‚«ã‚¦ãƒ³ãƒˆ
- アップãƒãƒ¼ãƒ‰ã™ã‚‹ãƒ•ã‚¡ã‚¤ãƒ«ã‚µã‚¤ã‚ºã®ä¿å˜
Ruby エンジニアã«ã¨ã£ã¦ã¯ Dataflow ç‰ã®å¤§è¦æ¨¡ãƒ‡ãƒ¼ã‚¿å‡¦ç†ãƒ„ールã¨æ¯”ã¹ã¦å¦ç¿’コストãŒä½Žã„ãŸã‚ã€å°Žå…¥ã‚’比較的容易ã«è¡Œã†ã“ã¨ãŒã§ãã¾ã™ã€‚
コンセプト
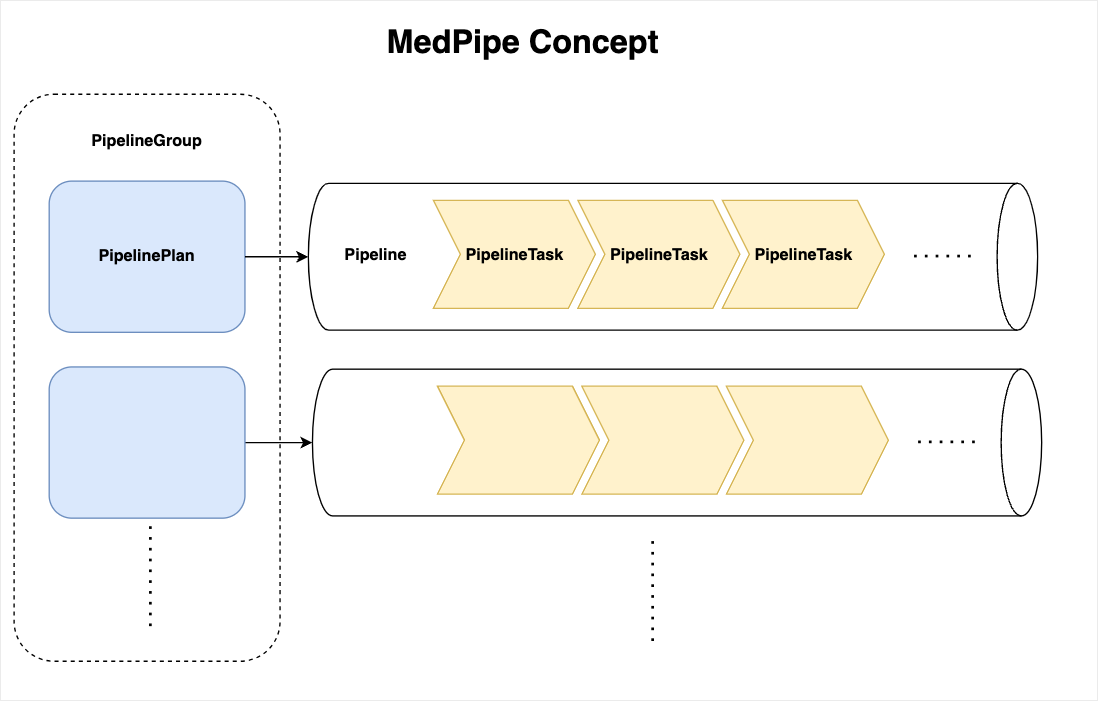
MedPipe ã§ã¯ Pipeline ã« PipelineTask を登録ã—ã€ãã‚Œã‚’é †ç•ªã«å®Ÿè¡Œã—ã¾ã™ã€‚
PipelineTask ã¯ã‚„ã‚ŠãŸã„ã“ã¨ãã®ã‚‚ã®ã§ã‚ã‚‹ãŸã‚ã€ç‹¬è‡ªã§å®Ÿè£…ã™ã‚‹å¿…è¦ãŒã‚ã‚Šã¾ã™ã€‚
PipelineTask ãŒå®Ÿè£…ã™ã‚‹å¿…è¦ã®ã‚るメソッド㯠call ã®ã¿ã§éžå¸¸ã«ã‚·ãƒ³ãƒ—ルã§ã™ã€‚
def call(context, prev_result) yield "次ã®Taskã®ç¬¬äºŒå¼•æ•°ã«æ¸¡ã™å€¤" end
ãŸã ã—ã€å¤§é‡ã®ãƒ‡ãƒ¼ã‚¿ã‚’扱ã†éš›ã«ã¯å…¨éƒ¨ã®ãƒ‡ãƒ¼ã‚¿ã‚’メモリã«ã®ã›ã¦æ¬¡ã® Task ã«æ¸¡ã™ã‚ã‘ã«ã¯ã„ãã¾ã›ã‚“。
ãã“ã§ã€åŸºæœ¬çš„ã«ã¯ Enumerable::Lazy を後続 Task ã«æ¸¡ã—ã¾ã™ã€‚
(lazy 㧠Enumerable ã‚’ Enumerable::Lazy ã«å¤‰æ›ã§ãã¾ã™)
例
def call(_context, _) yield HogeLog.find_each.lazy end
後続 Task 㯠Enumerable::Lazy ã‚’å—ã‘å–ã‚Šã€map ã§å‡¦ç†ã‚’挟むã“ã¨ã§ Enumerable::Lazy ã‚’ç¶æŒã§ãã¾ã™ã€‚
def call(_context, records) yield records.map { |record| format_line(record) } end
PipelineTask ã®ä»–ã«ã‚‚ PipelineTask ã‚’ Pipeline ã«ç™»éŒ²ã™ã‚‹å‡¦ç†ãªã©ä½¿ã†æº–å‚™ã¯å¿…è¦ã§ã™ã€‚
Usageã¨ã‚µãƒ³ãƒ—ルをå‚考ã«ã—ã¦ãã ã•ã„。
DB ã‹ã‚‰ã®ãƒ‡ãƒ¼ã‚¿å–得方法
実務㧠find_each を使ã†å ´åˆã«ã¯ 2 ã¤ã®å•é¡ŒãŒã‚ã‚Šã¾ã—ãŸã€‚
- ActiveRecord ã®ãƒ¡ãƒ¢ãƒªä½¿ç”¨é‡ãŒå¤šã„
- クエリãŒæœ€é©åŒ–ã•ã‚Œãªã„
1 ã«é–¢ã—ã¦ã¯ in_batches + pluck を使ã†ã“ã¨ã§è§£æ±ºã§ãã¾ã™ãŒã€2 ã«é–¢ã—ã¦ã¯è§£æ±ºã§ãã¾ã›ã‚“。
å‚考: Railsã§in_batches使ã†ã¨ã¨ã¦ã‚‚é…ã„
ã“れを解決ã™ã‚‹ãŸã‚ã«ã€MedPipe ã§ã¯ BatchReader ã¨ã„ã†ã‚¯ãƒ©ã‚¹ã‚’開発ã—ã¾ã—ãŸã€‚
使用例:
def call(_context, _) yield MedPipe::BatchReader.new( HogeLog, scope: HogeLog.where(created_at: @target_date.all_day), pluck_columns:, batch_size: BATCH_SIZE ).each.lazy end
ã“ã‚Œã«ã‚ˆã£ã¦ find_each ã®ã‚ˆã†ã«1件ãšã¤ã€pluck_columns 㧠pluck ã•ã‚ŒãŸãƒ‡ãƒ¼ã‚¿ã‚’後続 Task ã«æ¸¡ã™ã“ã¨ãŒã§ãã¾ã™ã€‚
プãƒãƒ•ã‚¡ã‚¤ãƒªãƒ³ã‚°ã®ä»•æ–¹
実務ã§ã¯ memory_profiler を用ã„ã¦ã€ä»¥ä¸‹ã®ã‚ˆã†ãªã‚³ãƒ¼ãƒ‰ã§ãƒ—ãƒãƒ•ã‚¡ã‚¤ãƒªãƒ³ã‚°ã‚’è¡Œã„ã¾ã—ãŸã€‚ ※ 執ç†ã«ã‚ãŸã‚Šä¸€éƒ¨ä¿®æ£ã—ã¦ã„ã¾ã™ã€‚
module Profiler class << self ... def report(&block) start_time = Time.current result = MemoryProfiler.report(&block) elapsed_time = Time.current - start_time puts "\n\n===== Profiler Report =====" puts "Total allocated: #{bytes_to_mb(result.total_allocated_memsize)} MB (#{result.total_allocated} objects)" puts "Total retained: #{bytes_to_mb(result.total_retained_memsize)} MB (#{result.total_retained} objects)" puts "Elapsed time: #{elapsed_time.round(2)} sec" end ... private ... # bytes to MB å°æ•°ç‚¹ç¬¬äºŒä½ã¾ã§ def bytes_to_mb(bytes) (bytes / 1024.0 / 1024.0).round(2) end end end
class PipelineTask::Profiler def call(_context, input) Profiler.report do # Lazy ã®å ´åˆã€æ¸¬å®šã™ã‚‹ãŸã‚ã«ç™ºç«ã™ã‚‹ input.force if input.is_a?(Enumerator::Lazy) yield(input) end end end
pipeline.apply(PipelineTask::Profiler.new)
æ—¢å˜ã®ã‚¹ã‚¯ãƒªãƒ—トを修æ£ã™ã‚‹ã“ã¨ãªãã€ãƒ—ãƒãƒ•ã‚¡ã‚¤ãƒªãƒ³ã‚°ã‚’è¡Œã†ã“ã¨ãŒã§ãã¾ã™ã€‚
ãŠã‚ã‚Šã«
本記事ã§ã¯ã€MedPipe ã®ç´¹ä»‹ã‚’è¡Œã„ã¾ã—ãŸã€‚本 gem ã¯å¼Šç¤¾åˆã®ã‚ªãƒ¼ãƒ—ンソース㮠gem ã§ã™ã€‚
普段様々㪠OSS ã®ãŠä¸–話ã«ãªã£ã¦ã„ã‚‹ãŸã‚ã€æä¾›ã™ã‚‹å´ã¨ã—ã¦æ¥ç•Œã«è²¢çŒ®ã§ãã‚‹ã“ã¨ã‚’嬉ã—ãæ€ã„ã¾ã™ã€‚
OSS ã¨ã—ã¦ä¸–ã«å‡ºã™ã“ã¨ã‚’許å¯ã—ã¦ã„ãŸã ã„ãŸä¼šç¤¾ã‚„一緒ã«é–‹ç™ºã—ãŸåŒåƒšã®è¿‘è—¤ã•ã‚“(@tetetratra)ã«æ„Ÿè¬ã§ã™!
実装ãŒå‚考ã«ãªã£ãŸã‚Šã€ä½¿ã£ã¦ã¿ã¦ã‚ˆã‹ã£ãŸå ´åˆã¯ã€ãœã² MedPipe ã® GitHub リãƒã‚¸ãƒˆãƒªã«ã‚¹ã‚¿ãƒ¼ã‚’ã„ãŸã ã‘ã‚‹ã¨åŠ±ã¿ã«ãªã‚Šã¾ã™ã€‚
是éžèªè€…ã«ãªã£ã¦ãã ã•ã„ï¼
メドピアã§ã¯ä¸€ç·’ã«åƒã仲間を募集ã—ã¦ã„ã¾ã™ã€‚ ã”応募をãŠå¾…ã¡ã—ã¦ãŠã‚Šã¾ã™ï¼
■募集ãƒã‚¸ã‚·ãƒ§ãƒ³ã¯ã“ã¡ã‚‰ medpeer.co.jp
■エンジニア紹介ページã¯ã“ã¡ã‚‰ engineer.medpeer.co.jp
■メドピア公å¼YouTube  www.youtube.com
■メドピア公å¼note
style.medpeer.co.jp