パーティクルフィルタ/粒子フィルタ(Particle filter)、逐次モンテカルロ法(Sequential Monte Carlo: SMC)など様々な呼び方がありますが、この記事ではパーティクルフィルタという呼び方を使います。このパーティクルフィルタをPythonで実装して状態空間モデルの潜在変数を推定することを試したいと思います。
状態空間モデルにはさらに細かくいくつものモデルが分かれていますが、今回はシンプルなモデルであるローカルモデル(1階差分トレンドモデル)を対象として扱います。この状態空間モデルのトレンドの種類に他に何があるかを知りたい場合はココをご参考としてください。
\begin{aligned}
x_t &= x_{t-1} + v_t, \quad v_t \sim N(0, \alpha^2\sigma^2)\quad &\cdots\ {\rm System\ model} \\
y_t &= x_{t} + w_t, \quad w_t \sim N(0, \sigma^2)\quad &\cdots\ {\rm Observation\ model}
\end{aligned}
$x_t$は時刻tにおける潜在状態を表し、$y_t$は観測値を表します。
利用するデータは「予測に生かす統計モデリングの基本」のサンプルデータ(http://daweb.ism.ac.jp/yosoku/) を使います。
 このテストデータはこちら(http://daweb.ism.ac.jp/yosoku/) から入手できます。
このテストデータはこちら(http://daweb.ism.ac.jp/yosoku/) から入手できます。
パーティクルフィルタを使うと、下記のグラフのように、各時点での潜在状態をパーティクル、つまり乱数を用いて推定することができます。赤い粒々がパーティクルで、この粒々に尤度をベースにした重みを割り当てて、加重平均をとることでフィルタリングを行うことができ、緑色の線で表しているような潜在状態を推定することができます。観測モデル(Observation model)が表しているように青色の観測値(いま手に入れているデータ)はこの潜在状態から正規分布に従うようなノイズが発生しており、[潜在状態の値] + [ノイズ] =[実現値としての観測値] となっていることを仮定しているモデルとなります。

以下、このパーティクルフィルタでフィルタリングを行い、潜在状態の値を推定するアルゴリズムを説明します。
1. Pythonコード
コアとなるParticleFilterのクラスは下記です。
コードの全文はコチラ
class ParticleFilter(object):
def __init__(self, y, n_particle, sigma_2, alpha_2):
self.y = y
self.n_particle = n_particle
self.sigma_2 = sigma_2
self.alpha_2 = alpha_2
self.log_likelihood = -np.inf
def norm_likelihood(self, y, x, s2):
return (np.sqrt(2*np.pi*s2))**(-1) * np.exp(-(y-x)**2/(2*s2))
def F_inv(self, w_cumsum, idx, u):
if np.any(w_cumsum < u) == False:
return 0
k = np.max(idx[w_cumsum < u])
return k+1
def resampling(self, weights):
w_cumsum = np.cumsum(weights)
idx = np.asanyarray(range(self.n_particle))
k_list = np.zeros(self.n_particle, dtype=np.int32) # サンプリングしたkのリスト格納場所
# 一様分布から重みに応じてリサンプリングする添え字を取得
for i, u in enumerate(rd.uniform(0, 1, size=self.n_particle)):
k = self.F_inv(w_cumsum, idx, u)
k_list[i] = k
return k_list
def resampling2(self, weights):
"""
計算量の少ない層化サンプリング
"""
idx = np.asanyarray(range(self.n_particle))
u0 = rd.uniform(0, 1/self.n_particle)
u = [1/self.n_particle*i + u0 for i in range(self.n_particle)]
w_cumsum = np.cumsum(weights)
k = np.asanyarray([self.F_inv(w_cumsum, idx, val) for val in u])
return k
def simulate(self, seed=71):
rd.seed(seed)
# 時系列データ数
T = len(self.y)
# 潜在変数
x = np.zeros((T+1, self.n_particle))
x_resampled = np.zeros((T+1, self.n_particle))
# 潜在変数の初期値
initial_x = rd.normal(0, 1, size=self.n_particle) #--- (1)
x_resampled[0] = initial_x
x[0] = initial_x
# 重み
w = np.zeros((T, self.n_particle))
w_normed = np.zeros((T, self.n_particle))
l = np.zeros(T) # 時刻毎の尤度
for t in range(T):
print("\r calculating... t={}".format(t), end="")
for i in range(self.n_particle):
# 1階差分トレンドを適用
v = rd.normal(0, np.sqrt(self.alpha_2*self.sigma_2)) # System Noise #--- (2)
x[t+1, i] = x_resampled[t, i] + v # システムノイズの付加
w[t, i] = self.norm_likelihood(self.y[t], x[t+1, i], self.sigma_2) # y[t]に対する各粒子の尤度
w_normed[t] = w[t]/np.sum(w[t]) # 規格化
l[t] = np.log(np.sum(w[t])) # 各時刻対数尤度
# Resampling
#k = self.resampling(w_normed[t]) # リリサンプリングで取得した粒子の添字
k = self.resampling2(w_normed[t]) # リリサンプリングで取得した粒子の添字(層化サンプリング)
x_resampled[t+1] = x[t+1, k]
# 全体の対数尤度
self.log_likelihood = np.sum(l) - T*np.log(n_particle)
self.x = x
self.x_resampled = x_resampled
self.w = w
self.w_normed = w_normed
self.l = l
def get_filtered_value(self):
"""
尤度の重みで加重平均した値でフィルタリングされ値を算出
"""
return np.diag(np.dot(self.w_normed, self.x[1:].T))
def draw_graph(self):
# グラフ描画
T = len(self.y)
plt.figure(figsize=(16,8))
plt.plot(range(T), self.y)
plt.plot(self.get_filtered_value(), "g")
for t in range(T):
plt.scatter(np.ones(self.n_particle)*t, self.x[t], color="r", s=2, alpha=0.1)
plt.title("sigma^2={0}, alpha^2={1}, log likelihood={2:.3f}".format(self.sigma_2,
self.alpha_2,
self.log_likelihood))
plt.show()
2. パーティクルフィルタの仕組み
以下、1フレームずつ解説します。
STEP1:
まずt-1時点から始まりますが、t=1の場合は初期値が必要です。ここでは、初期値に$N(0, 1)$に従う乱数を生成してこれを初期値することとします。t>1の場合は一つ前の時刻の結果がこの$X_{re,t-1|t-1}$にあたります。
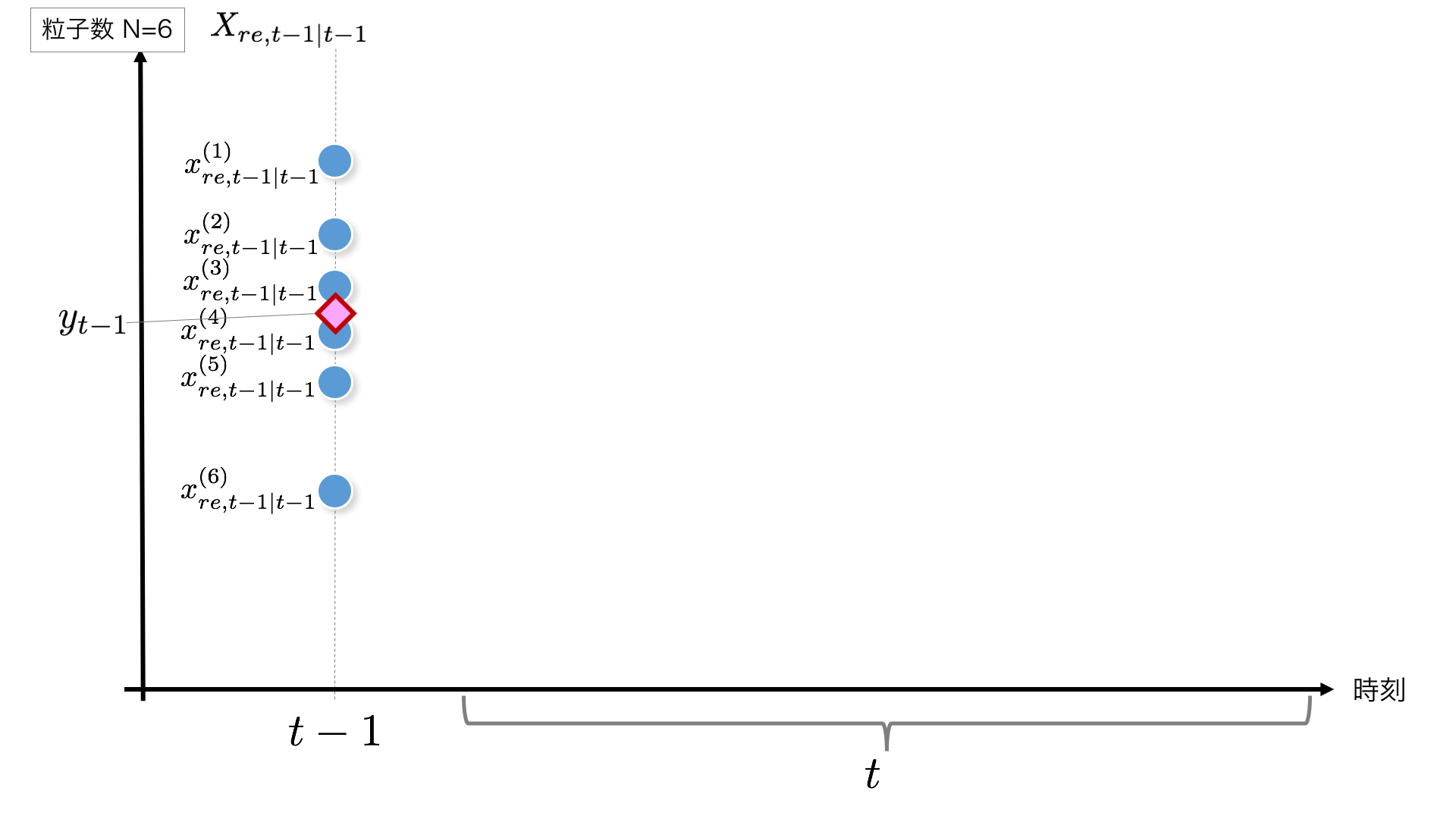
粒子の初期化部分のコードはこちらです。
initial_x = rd.normal(0, 1, size=self.n_particle) #--- (1)
STEP2:
システムモデルに従い、いまの時刻$t-1$から次の時刻に$t$以降する際、ノイズが乗ります。ここでは正規分布$N(0, \alpha^2\sigma^2)$に従うことを想定しています。これを粒子の数だけ実施します。
x_t = x_{t-1} + v_t, \quad v_t \sim N(0, \alpha^2\sigma^2)\quad \cdots\ {\rm System\ model}

コードでは下記のように各粒子に$N(0, \alpha^2\sigma^2)$に従うノイズを加えています。
v = rd.normal(0, np.sqrt(self.alpha_2*self.sigma_2)) # System Noise #--- (2)
x[t+1, i] = x_resampled[t, i] + v # システムノイズの付加
STEP3:
ノイズが乗った後の粒子の値 $x_{t|t-1}$が全て得られたあと、時刻$t$の観測値である$y_t$が得られるので、各粒子の尤度を計算します。
ここで計算される尤度は、いま得られた観測値$y_t$に対する粒子の最もらしさですね。これを粒子の重みとして利用したいので、変数に入れてとっておきます。この尤度計算も粒子の数だけ実行します。
尤度は
p(y_t|x_{t|t-1}^{(i)}) = w_{t}^{(i)} = {1 \over \sqrt{2\pi\sigma^2}} \exp \left\{
-{1 \over 2} {(y_t - x_{t|t-1}^{(i)})^2 \over \sigma^2}
\right\}
で計算します。
全ての粒子で尤度を計算した後、正規化した重み(全部足して1になるように)
\tilde{w}^{(i)}_t = { w^{(i)}_t \over \sum_{i=1}^N w^{(i)}_t}
も用意しておきます。

def norm_likelihood(self, y, x, s2):
return (np.sqrt(2*np.pi*s2))**(-1) * np.exp(-(y-x)**2/(2*s2))
w[t, i] = self.norm_likelihood(self.y[t], x[t+1, i], self.sigma_2) # y[t]に対する各粒子の尤度
w_normed[t] = w[t]/np.sum(w[t]) # 規格化
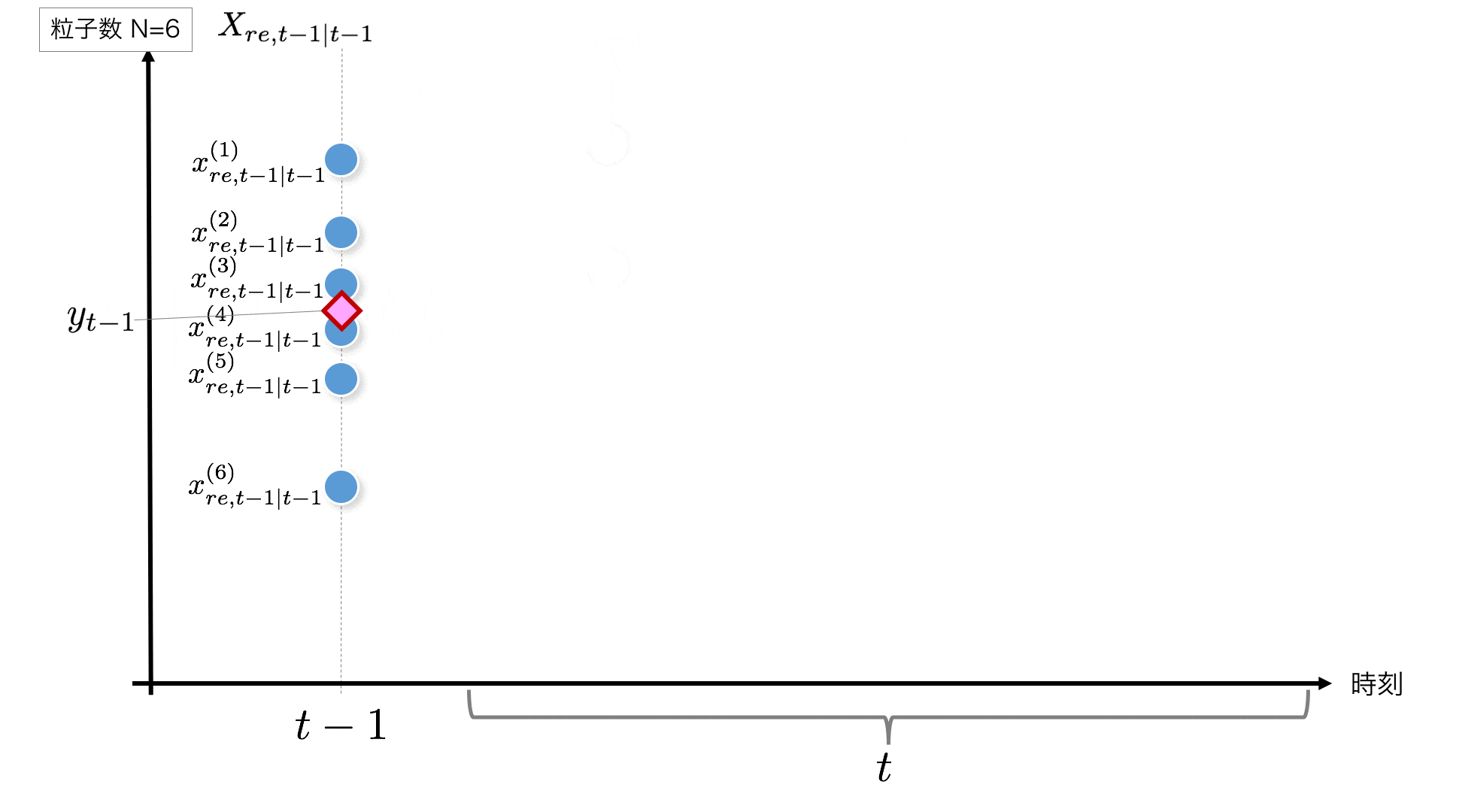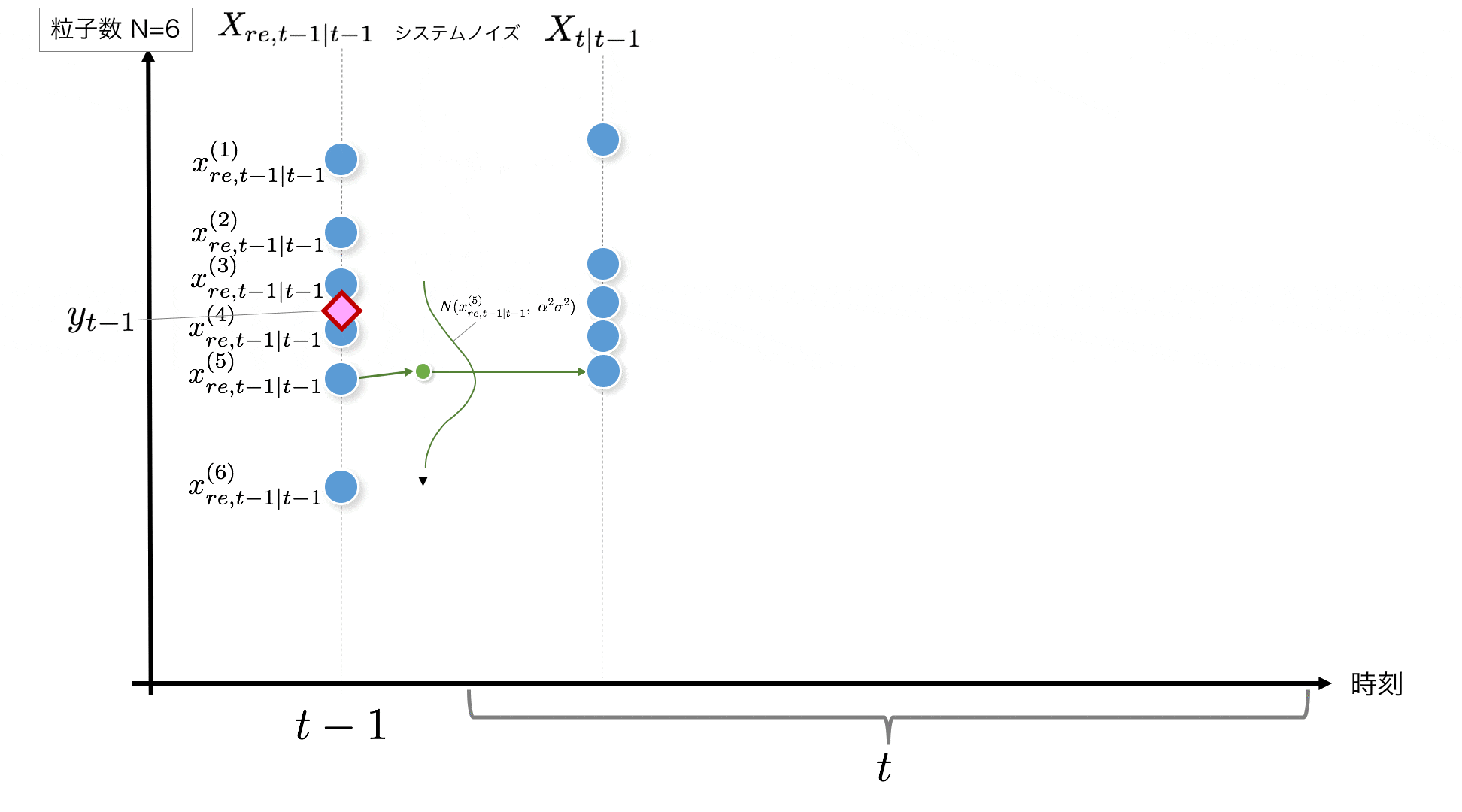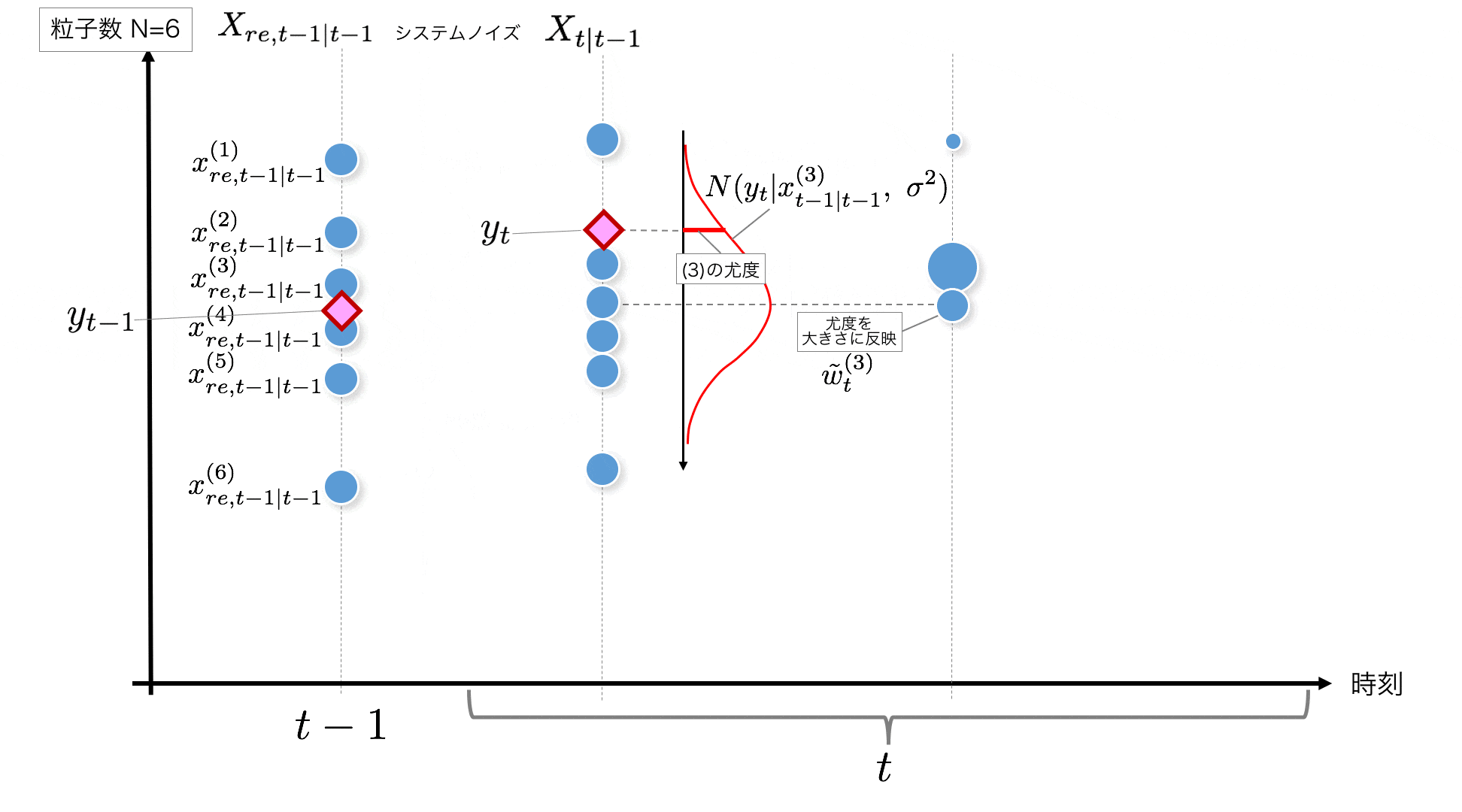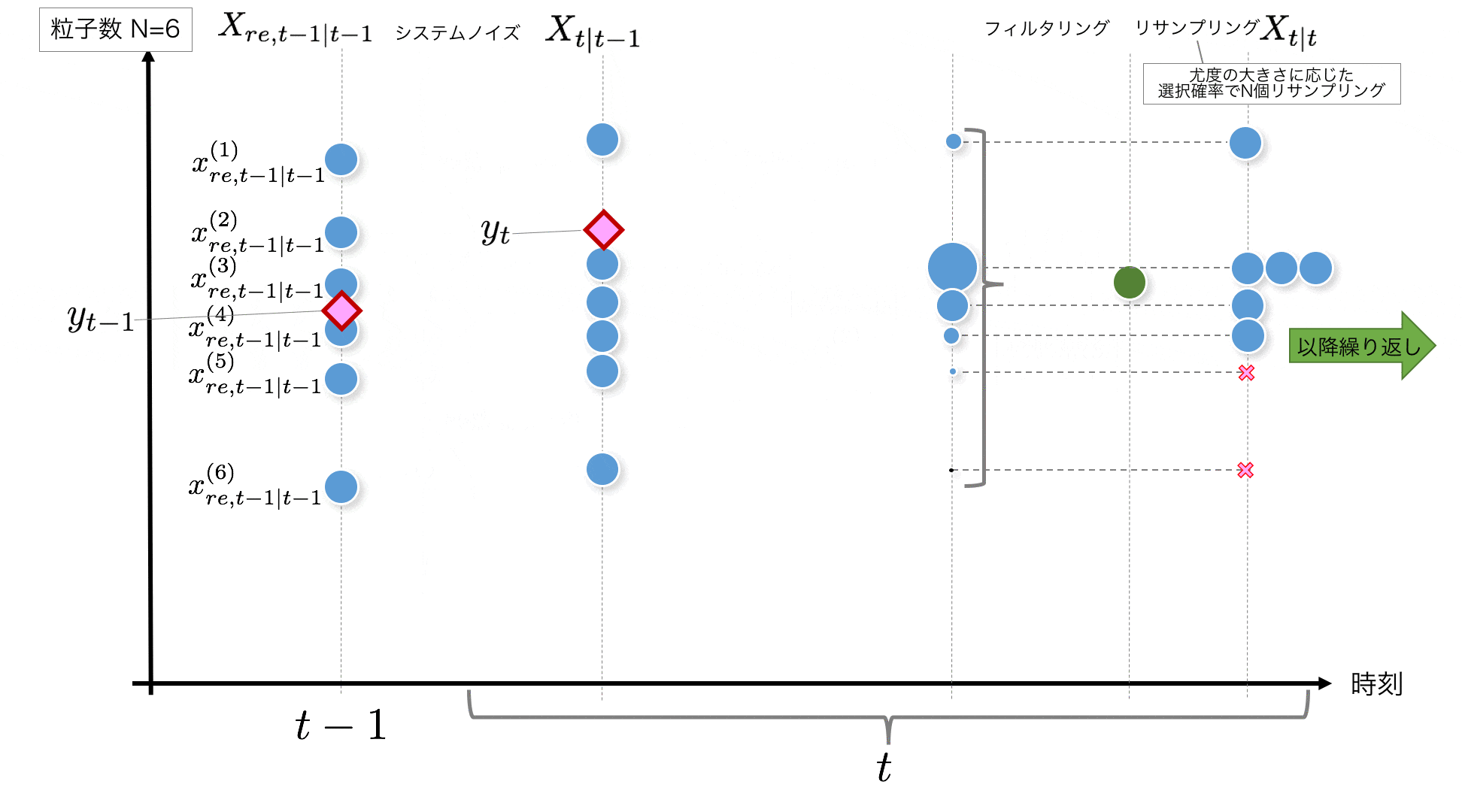
STEP4:
先ほどとっておいた重み$\tilde{w}^{(i)}_t$を使って加重平均
\sum_{i=1}^N \tilde{w}^{(i)}_t x_{t|t-1}^{(i)}
を計算して、フィルタリングした$x$の値とします。

コードでは下記のget_filtered_value()が該当します。
def get_filtered_value(self):
"""
尤度の重みで加重平均した値でフィルタリングされ値を算出
"""
return np.diag(np.dot(self.w_normed, self.x[1:].T))
STEP5:
尤度に基づく重み $\tilde{w}^{(i)}_t$ を抽出される確率とみなして、
X_{t|t-1}=\{x_{t|t-1}^{(i)}\}_{i=1}^N
からN個サンプルし直します。これはリサンプリングと言われますが、尤度の低い粒子は消滅させてしまい、尤度の高い粒子を複数個に分裂するような操作のイメージです。

k = self.resampling2(w_normed[t]) # リサンプリングで取得した粒子の添字(層化サンプリング)
x_resampled[t+1] = x[t+1, k]
リサンプリングするときは、下記のように経験分布関数を$\tilde{w}^{(i)}_t$ から作成し、その逆関数を作ることで、(0,1]の範囲の一様乱数から、重みに準じた確率で各粒子を取り出すことができます。

F_inv()が逆関数の処理をするコード、resampling2()が層化サンプリングを行うコードです。層化サンプリングは「カルマンフィルタ ―Rを使った時系列予測と状態空間モデル―(統計学One Point 2)」を参考に実装しています。
\begin{aligned}
u &\sim U(0, 1) \\
k_i &= F^{-1}((i-1+u)/N) \quad i=1,2,\cdots, N \\
x^{(i)}_{t|t} &= x^{(k_i)}_{t|t-1}
\end{aligned}
のような感じですね。(0, 1]の間の一様乱数をN個生成する代わりに(0, 1/N]の間の一様乱数1つだけ生成して、(0, 1]の間に1/Nづつずらして均等な幅でN個サンプリングしていることになっています。

def F_inv(self, w_cumsum, idx, u):
if np.any(w_cumsum < u) == False:
return 0
k = np.max(idx[w_cumsum < u])
return k+1
def resampling2(self, weights):
"""
計算量の少ない層化サンプリング
"""
idx = np.asanyarray(range(self.n_particle))
u0 = rd.uniform(0, 1/self.n_particle)
u = [1/self.n_particle*i + u0 for i in range(self.n_particle)]
w_cumsum = np.cumsum(weights)
k = np.asanyarray([self.F_inv(w_cumsum, idx, val) for val in u])
return k
この操作をt=1からTまで実施することで、粒子の生成とフィルタリングを行うことができます。
その結果が下記のグラフになります。

3. 実装が正しいか、Kalman Filterの結果と比べて検証する
さて、実装したパーティクルフィルタが正しい挙動をするか、念のためStatsModelsにあるカルマンフィルタの結果と突き合わせて同じような結果を出力しているかを確認したいと思います
# Unobserved Components Modeling (via Kalman Filter)の実行
import statsmodels.api as sm
# Fit a local level model
mod_ll = sm.tsa.UnobservedComponents(df.data.values, 'local level')
res_ll = mod_ll.fit()
print(res_ll.summary())
# Show a plot of the estimated level and trend component series
fig_ll = res_ll.plot_components(legend_loc="upper left", figsize=(12,8))
plt.tight_layout()

これを先ほどのパーティクルフィルタの結果と重ねてみると…、ほとんどぴったり合っていますね!![]()

以上、パーティクルフィルターの実装でした。なぜこれがうまくいくのかの解説は、予測に生かす統計モデリングの基本 樋口 知之(著)がとてもわかりやすいので必読です。
宿題
- 各時点の信頼区間の追加
- T時点以降の予測
- 固定ラグ平滑化をまだ入れていないので入れたい。
- ハイパーパラメーターサーチがGrid Searchでダサいので考えたい。
- ローカルモデル以外のトレンドや、Seasonal項の追加などに対応
- 高速化
参考
- 本ページで利用したコード
https://github.com/matsuken92/Qiita_Contents/blob/master/particle_filter/particle_filter_class.ipynb - 予測に生かす統計モデリングの基本 樋口 知之(著)
https://www.amazon.co.jp/dp/4061557955 - カルマンフィルタ ―Rを使った時系列予測と状態空間モデル―(統計学One Point 2) 野村 俊一(著)
https://www.amazon.co.jp/dp/4320112539 - 計算統計 2 マルコフ連鎖モンテカルロ法とその周辺 (統計科学のフロンティア 12) 伊庭 幸人(著), 種村 正美(著)
https://www.amazon.co.jp/dp/4000068520 - StatsModels: Unobserved Components Modeling
http://www.statsmodels.org/stable/generated/statsmodels.tsa.statespace.structural.UnobservedComponents.html#statsmodels.tsa.statespace.structural.UnobservedComponents