At the moment ld-tools is not working. Main reason: 1000 Genomes project deleted VCFs containing rsIDs. Secondary reason: Plotly visualization library heatmap functionality has been fundamentally changed. To bring ld-tools back to life requires an almost complete code rewrite. I don't have the time, energy or funding for that right now. Let this repository remain published: maybe I will return to development or someone will make a fork.
| Программа | Основная функциональность |
|---|---|
| ld_area | отбирает для каждого из запрашиваемых вариантов все варианты в пределах двух порогов: не ниже определённого LD и не дальше заданных фланков |
| ld_lite | выводит LD и дистанцию для двух вариантов |
| ld_triangle | строит матрицы значений LD всех возможных пар вариантов исходного набора |
Комбинации генотипов в популяции по-идее должны распределяться случайно - к этому прикладывают руку многочисленные кроссинговеры. Но существуют пары аллелей, встречающиеся явно чаще, чем должно давать случайное распределение. Это и есть неравновесие по сцеплению (LD). Причины сабжа:
- Популяция пока очень молодая, и достаточного количества кроссинговеров ещё не успело произойти.
- Близость новоиспечённых мутаций друг к другу.
- Комбинация выгодна организму.
Возможно, единственный подходящий источник данных для расчёта LD - таблицы популяционно-генетического проекта 1000 Genomes. Вот крохотный отрывок одного из этих гигантских файлов:
| CHROM | POS | ID | ... | HG00096 | HG00097 | HG00099 | ... |
|---|---|---|---|---|---|---|---|
| 1 | 10177 | rs367896724 | ... | 1│0 | 0│1 | 0│1 | ... |
| 1 | 10235 | rs540431307 | ... | 0│0 | 0│0 | 0│0 | ... |
| 1 | 10352 | rs555500075 | ... | 1│0 | 1│0 | 0│1 | ... |
| ... | ... | ... | ... | ... | ... | ... | ... |
Любая из таблиц содержит данные только одной хромосомы. CHROM, POS и ID, как вы уже сами догадались - хромосома, позиция и rsID (reference SNP ID) каждого варианта. Следующие 6 столбцов я пропустил, т.к. они не участвуют в калькуляции LD. Дальше сложнее и интереснее. HG00096, HG00097 и др. - это идентификаторы сэмплов, т.е. ДНК 2504 добровольцев, представляющих 26 популяций, в свою очередь, принадлежащих 5 суперпопуляциям (азиаты, европейцы и т.д.). Огромное море разделённых вертикальной чертой единичек и ноликов - это фазированные генотипы. Что в 1000G означает "генотип"? Всё просто: 0 - в данной позиции имеется референсный аллель (такой же, как в этой же точке общепринятого образцово-показательного генома), 1 - альтернативный. Теперь как понять "фазированные"? Значит, точно известно, что цифры левее черты принадлежат строго одному гомологу хромосомы индивида, а правее - другому. Раз есть такая определённость принадлежности аллелей гомологам, то позволительно оперировать аллельными сочетаниями - гаплотипами. В нашем примере для вариантов rs367896724 и rs540431307 можно разглядеть гаплотипы 1 + 0 и 0 + 0. Поиск LD двух вариантов начинается с подсчёта в пределах выбранного вами набора сэмплов частоты 1) каждого из двух генотипов первого варианта, 2) второго, 3) какого-то одного гаплотипа. Математически, неравновесие по сцеплению - это отклонение наблюдаемой частоты гаплотипа от ожидаемой. Калькулятор в составе ld-tools получает D (базовое значение LD) вот так:
d = htype_alt_alt_freq - var_1_alt_freq * var_2_alt_freq
Здесь D ищется по альтернативным аллелям. Можно, в принципе, и по референсным, ну или по обоим. Как видно по формуле, ожидаемая частота гаплотипа 1 + 1 - произведение частот генотипов 1. LD в более сложных, но чаще используемых величинах - D' и r2 - рассчитывается несколько хитрее. Формулы и комментарии к ним можете посмотреть в соответствующем бэкендном модуле тулкита.
До появления ld_tools уже существовали мощные, популярные, разрабатываемые крупными лабораториями решения: LDlink, Linkage Disequilibrium Calculator, Haploreg и др.. На сегодняшний день по ряду функциональных моментов ld_tools занимает более выигрышную позицию.
- Не нужно вручную загружать по одному файлу, программа сама сканирует всю исходную папку;
- Автоматически распознаётся столбец с идентификаторами вариантов каждой таблицы.
- Тщательно отсеиваются мультиаллельные варианты, в том числе такие коварные, как вставки с повторами разной длины.
- Вплоть до 8 исходных файлов могут обрабатываться параллельно.
- После подготовки кэша работу можно проводить полностью оффлайн. Никаких тормознутых API и 503-сюрпризов.
- Не возникает конфликт, если на вход подаются варианты разных хромосом.
- Возможность тонкой настройки каждого запуска с помощью агрументов командной строки.
- Сохранение в конечные файлы настроек их получения.
- Геномные координаты - только GRCh38. Нет характерной для биоинформатических тулзов путаницы по этому вопросу.
- Человеко-понятные имена переменных и подробные комментарии к коду. Делаю всё, чтобы проект пригождался в том числе и для изучения программирования в биоинформатике.
- Отличная производительность на неигровых ноутбуках. Тестировал на ASUS VivoBook X712FA с довольно хилым Intel Core i5-8265U.
- Техподдержка лично от создателя программы, без посылания на Stack Overflow.
- Хотя бы чуть-чуть ознакомьтесь с командной строкой.
- Установите сторонние Python-компоненты: необходимый для парсинга VCF-файлов pysam, конструктор табличных принтов tabulate и визуализатор plotly. Советую это делать посредством Conda. Не убирайте из команды
conda installномера версий, иначе будут проблемы совместимости.
conda install python=3.7 pysam=0.15.4 tabulate plotly
- Скачайте архив с программой.
- Распакуйте его в любое место.
- В терминале перейдите в разархивированную папку.
cd /path/to/ld-tools-master
- Выведите справку по интересующему компоненту:
python3 ld_triangle -h
- Любым компонентом этой программы будут проигнорированы:
- мультиаллельные варианты;
- варианты с не-rs-идентификаторами;
- варианты, ставшие известными науке уже после выхода последней версии 1000 Genomes.
- Лучше исключать из исходных выборок данные X и Y-хромосом по причине нерешённой разработчиками 1000 Genomes проблемы с PAR-регионами.
- Нельзя отключать интернет при подготовке данных, необходимых для быстрого оффлайн-вычисления LD. Этот процесс производится лишь однократно и занимает примерно полдня.
- Большие тепловые карты LD-значений, вроде 500х500 rsIDs, могут попросту не прорисоваться вашим браузером.
Каждый исходный вариант порождает отдельный файл надпорогово-сцепленных с ним вариантов. Идея программы навеяна ныне заброшенным онлайн-инструментом HaploReg.
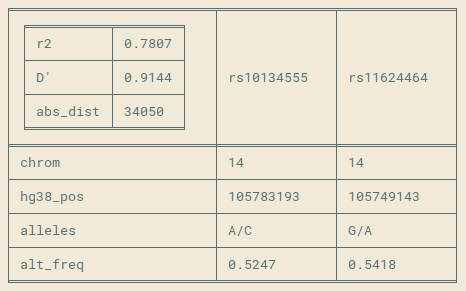
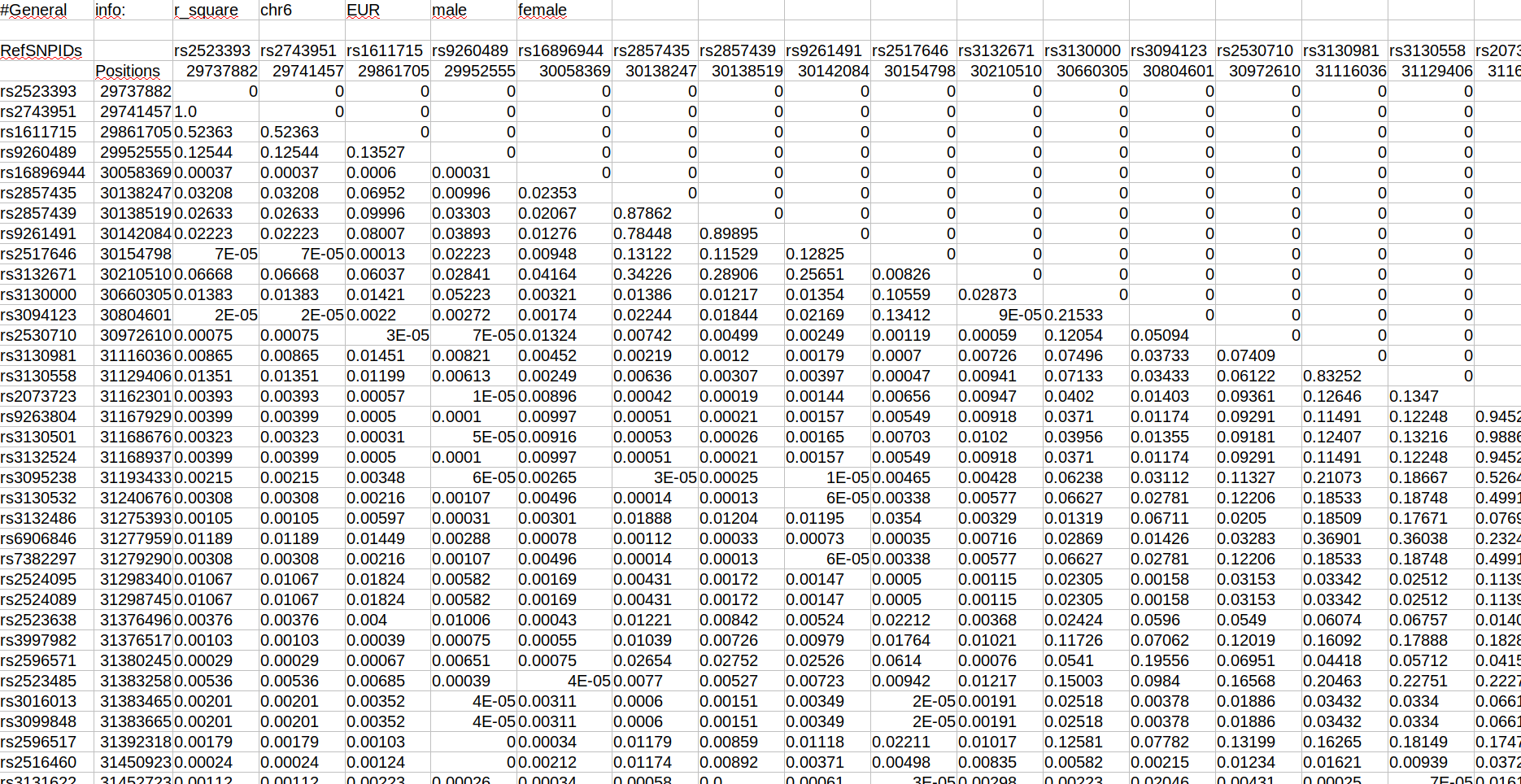
- TSV. Формат по умолчанию. Это - обычные таблицы, удобные и информативные для клиницистов, в то же время, легко обрабатываемые скриптами. Ключевые столбцы - r2, D' и дистанция между найденными и запрашиваемой мутациями. Полезное дополнение - базовые аннотации каждой мутации (в том числе запрашиваемой): позиция, идентификатор, референсный и альтернативный аллели, тип, а также частота альтернативного аллеля.
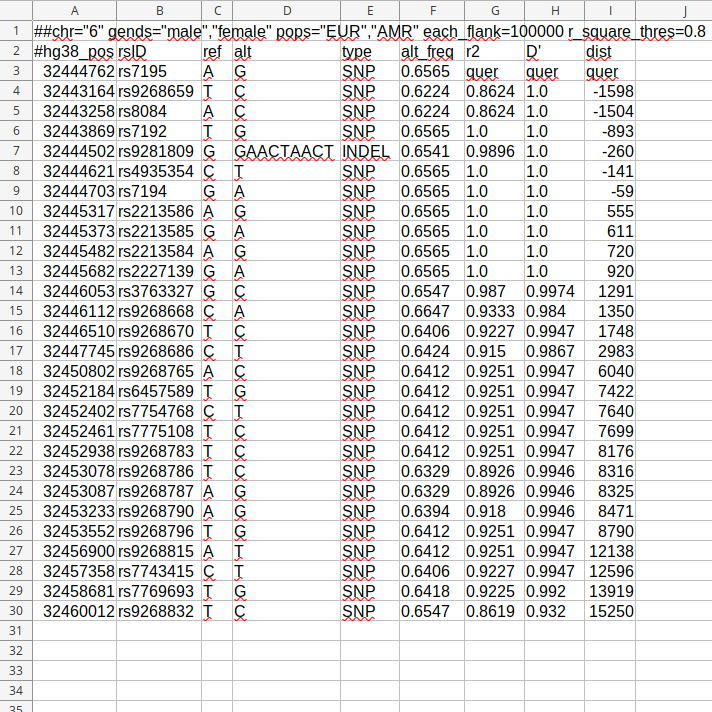
- JSON. По содержанию совпадает с TSV, но больше ориентирован на программистов.
- Набор одних лишь rsIDs. Такая разновидность вывода лучше всего подходит для дальнейшего аннотирования мутаций специализированными программами (high_perf_bio, Ensembl VEP и др.).
Для работы лишь с двумя вариантами. Пара подаётся в качестве аргументов команды. Конечная таблица отображается здесь же, в терминале. Дополняющий основные результаты набор аннотаций аналогичен тому, что выводит ld_area.
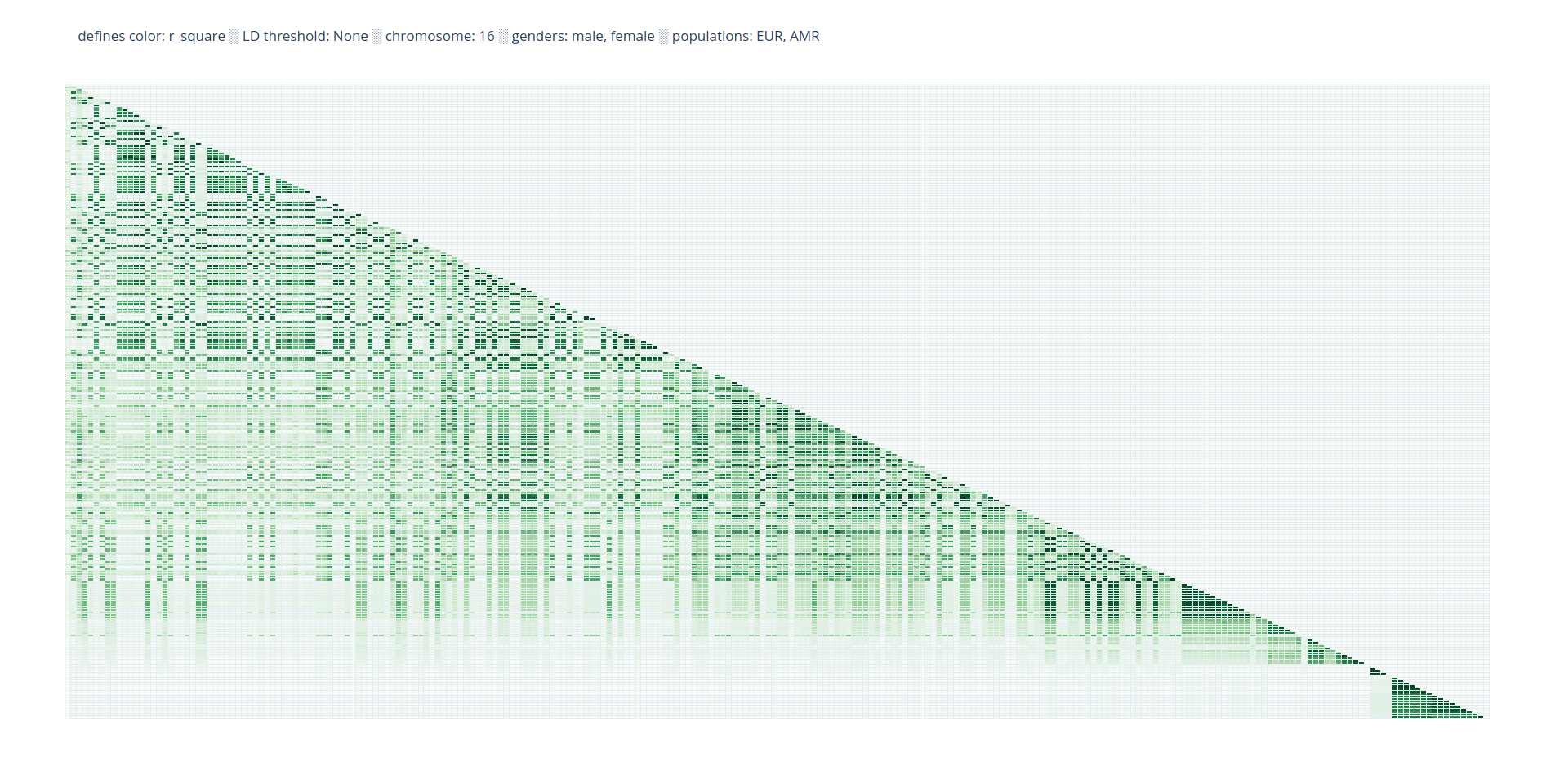
Каждая матрица создаётся по всем парным сочетаниям соответствующего единого набора rsIDs. Важно отметить, что если уже найдено LD для пары вариант_x-вариант_y, то обработка пары вариант_y-вариант_x допущена не будет. Поэтому вместо квадратов с результатами получаются прямоугольные треугольники. Программа является написанной с нуля десктопной реализацией идеи компонента LDmatrix веб-сервиса LDlink.
Подобрал такие палитры Plotly, с которыми LD-карты не будут смотреться вырвиглазно.
algae, amp, blues, blugrn, bluyl, brwnyl, bugn, bupu, burg, burgyl, darkmint, deep, dense, emrld, gnbu, greens, greys, magenta, matter, mint, oranges, orrd, oryel, peach, pinkyl, pubu, pubugn, purd, purp, purples, purpor, rdpu, redor, reds, speed, sunset, sunsetdark, teal, tealgrn, tempo, turbid, ylgn, ylgnbu, ylorbr, ylorrd
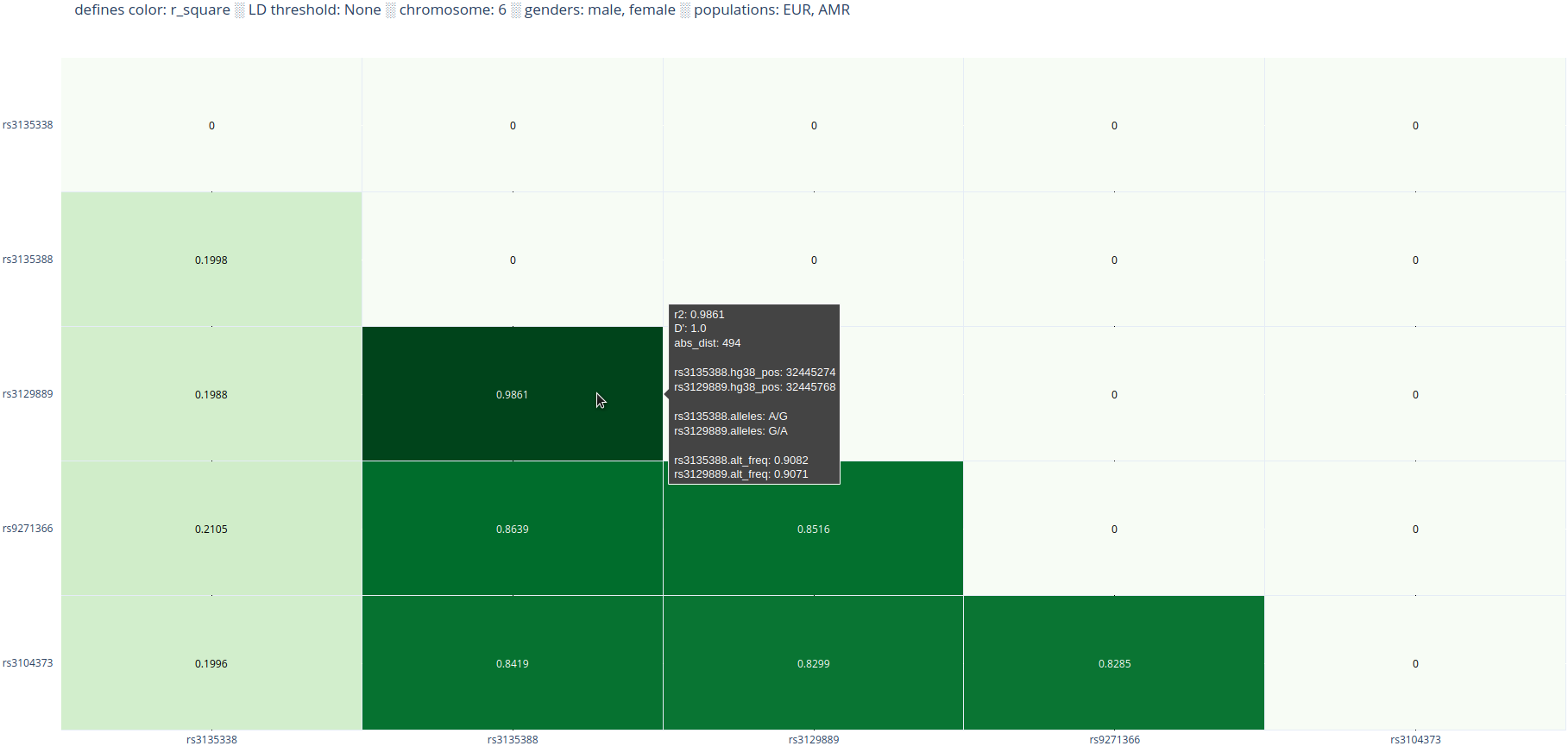
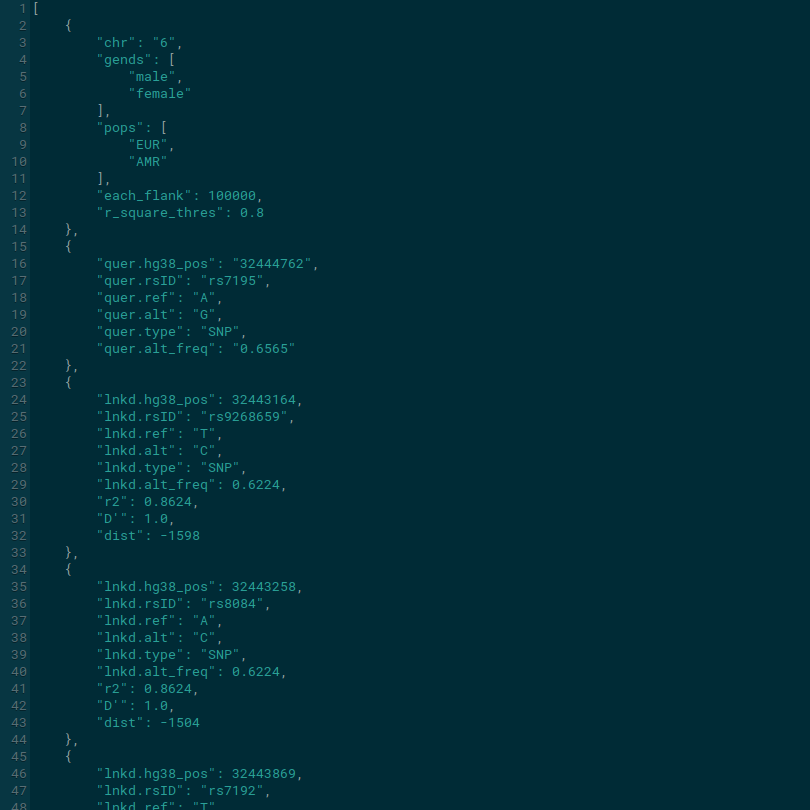
Основной тип вывода ld_triangle — тепловые карты (таблицы — лишь полезное дополнение), поэтому речь далее будет идти, в основном, о визуализированных результатах.
На пересечении варианта по оси X и варианта по оси Y отображается вычисленное для них LD. Оно может быть представлено в виде вещественного числа, степени насыщенности окраски квадратика, либо сочетанием одного и другого.
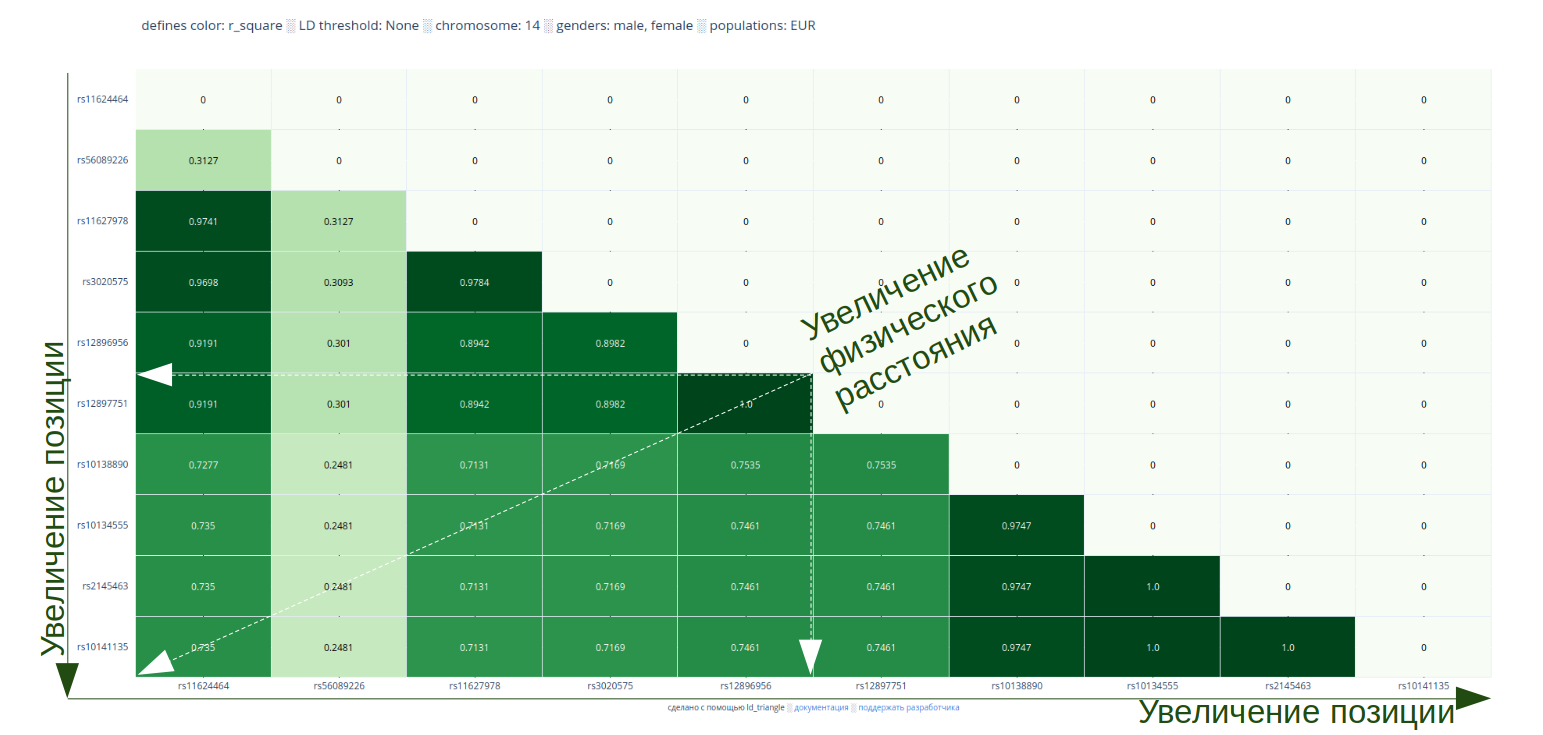 Программа сортирует варианты по возрастанию их позиции в хромосоме. Это позволяет упорядочить результаты по физическому расстоянию внутри пар. В LD-матрице rsID-лейблы располагаются таким образом, что по Y увеличение позиции вариантов идёт сверху вниз, а по X — слева направо. Исходя из этого, относительно любого квадратика, дистанция между вариантами, образующими остальные квадратики, увеличивается влево, вниз и по "результирующей" диагонали (см. рисунок выше). Следовательно, самая маленькая дистанция будет у пар вариантов на гипотенузе, а самая большая — около пересечения катетов.
Программа сортирует варианты по возрастанию их позиции в хромосоме. Это позволяет упорядочить результаты по физическому расстоянию внутри пар. В LD-матрице rsID-лейблы располагаются таким образом, что по Y увеличение позиции вариантов идёт сверху вниз, а по X — слева направо. Исходя из этого, относительно любого квадратика, дистанция между вариантами, образующими остальные квадратики, увеличивается влево, вниз и по "результирующей" диагонали (см. рисунок выше). Следовательно, самая маленькая дистанция будет у пар вариантов на гипотенузе, а самая большая — около пересечения катетов.
Другие особенности.
-
Можно не закрашивать клеточки с теми значениями LD, которые ниже определённого порога. Тогда при рассмотрении матрицы будет проще сосредоточиться на наиболее сильно сцепленных парах.

Делать небольшие полуоффтопные тьюториалы - это уже традиция для моих ридми:). ld_triangle использует библиотеку визуализации plotly, поэтому считаю нужным немного разобрать именно её. Это будет не огромная лавина отрывков кода для бездумного копирования, что привычно видеть на отечественных IT-порталах, а попытка лаконично акцентировать внимание на общем принципе. Поняв его, вы станете готовить графики без мучительного гугления.
Самый главный тезис - это что любая диаграмма plotly строится по экземпляру класса Figure. Он состоит из вложенных друг в друга объектов со свойствами обычных питоновских словарей и списков. Если у вас есть хотя бы базовые знания Python, вы можете легко читать и редактировать Figure-объект. К тому же, понимание его строения позволяет быстро ориентироваться в длиннющей простыне официальной документации.
Сохранить целиком объект тепловой карты попарных LD-значений можно флагом -j или --heatmap-json программы ld_triangle. Приводимые в примерах пути заменяйте на свои.
cd $HOME/Биоинформатика/ld-tools-master
python ld_triangle.py -S $HOME/Биоинформатика/SNP_data -D $HOME/Биоинформатика/1000_Genomes_data -f -e eur -j
Может показаться странным, что содержимое JSON гораздо объёмнее принта Figure-объекта. Для начала рассмотрим различия между выводом и файлом.
Компонент экземпляра класса Figure |
JSON | |
|---|---|---|
| Данные, формирующие базовую часть диаграммы | выводятся целиком | представлены целиком |
| Данные всплывающих текстов | выводятся целиком | представлены целиком |
| Настройки диаграммы | выводятся все изменённые и некоторые дефолтные | представлены и те, и другие |
Вытащим объект диаграммы обратно из JSON и выведем на экран.
python3
from plotly.io import read_json
ld_heatmap = read_json('/home/yourname/Биоинформатика/результаты/SNPs_LD_matr/chr6_r_SNPs.json')
print(ld_heatmap)
Перед нами он в урезанном виде. Но, на самом деле, в JSON-файле и в оперативной памяти мы имеем дело с одним и тем же объектом. Докажем это, построив по извлечённому из JSON объекту тепловую карту. Она получится такой же, как и изготовленная до этого полноценной программой.
ld_heatmap.show()
Очевидно, что отбор только наиболее значимых компонентов Figure-объекта делается для значительного повышения читабельности принта.
Теперь детально разберём выводимый фрагмент подкапотной части plotly-диаграммы. Как ранее говорилось в таблице, он состоит из самих данных и соответствующих метаданных, отредактированных ld_triangle настроек и небольшого количества настроек по умолчанию. Для простоты я сделал тепловую карту всего лишь по трём вариантам.
Figure({
'data': [{'colorscale': [[0.0, 'rgb(247,252,245)'], [0.125,
'rgb(229,245,224)'], [0.25, 'rgb(199,233,192)'],
[0.375, 'rgb(161,217,155)'], [0.5,
'rgb(116,196,118)'], [0.625, 'rgb(65,171,93)'], [0.75,
'rgb(35,139,69)'], [0.875, 'rgb(0,109,44)'], [1.0,
'rgb(0,68,27)']],
'hoverinfo': 'text',
'hovertext': [[0, 0, 0], ["\nr2: 0.0003<br>\nD':
0.0247<br>\nabs_dist: 1060331<br><br>\nrs1521.hg38_pos:
31382927<br>\nrs8084.hg38_pos:
32443258<br><br>\nrs1521.alleles:
C/T<br>\nrs8084.alleles: A/C<br><br>\nrs1521.alt_freq:
0.7376<br>\nrs8084.alt_freq: 0.5865\n", 0, 0], ["\nr2:
0.0027<br>\nD': 0.0668<br>\nabs_dist:
1060942<br><br>\nrs1521.hg38_pos:
31382927<br>\nrs7192.hg38_pos:
32443869<br><br>\nrs1521.alleles:
C/T<br>\nrs7192.alleles: T/G<br><br>\nrs1521.alt_freq:
0.7376<br>\nrs7192.alt_freq: 0.6332\n", "\nr2:
0.8216<br>\nD': 1.0<br>\nabs_dist:
611<br><br>\nrs8084.hg38_pos:
32443258<br>\nrs7192.hg38_pos:
32443869<br><br>\nrs8084.alleles:
A/C<br>\nrs7192.alleles: T/G<br><br>\nrs8084.alt_freq:
0.5865<br>\nrs7192.alt_freq: 0.6332\n", 0]],
'reversescale': False,
'showscale': False,
'type': 'heatmap',
'x': [rs1521, rs8084, rs7192],
'xgap': 1,
'y': [rs1521, rs8084, rs7192],
'ygap': 1,
'z': [[0, 0, 0], [0.0003, 0, 0], [0.0027, 0.8216, 0]]}],
'layout': {'annotations': [{'font': {'color': '#000000'},
'showarrow': False,
'text': '0',
'x': 'rs1521',
'xref': 'x',
'y': 'rs1521',
'yref': 'y'},
{'font': {'color': '#000000'},
'showarrow': False,
'text': '0',
'x': 'rs8084',
'xref': 'x',
'y': 'rs1521',
'yref': 'y'},
{'font': {'color': '#000000'},
'showarrow': False,
'text': '0',
'x': 'rs7192',
'xref': 'x',
'y': 'rs1521',
'yref': 'y'},
{'font': {'color': '#000000'},
'showarrow': False,
'text': '0.0003',
'x': 'rs1521',
'xref': 'x',
'y': 'rs8084',
'yref': 'y'},
{'font': {'color': '#000000'},
'showarrow': False,
'text': '0',
'x': 'rs8084',
'xref': 'x',
'y': 'rs8084',
'yref': 'y'},
{'font': {'color': '#000000'},
'showarrow': False,
'text': '0',
'x': 'rs7192',
'xref': 'x',
'y': 'rs8084',
'yref': 'y'},
{'font': {'color': '#000000'},
'showarrow': False,
'text': '0.0027',
'x': 'rs1521',
'xref': 'x',
'y': 'rs7192',
'yref': 'y'},
{'font': {'color': '#FFFFFF'},
'showarrow': False,
'text': '0.8216',
'x': 'rs8084',
'xref': 'x',
'y': 'rs7192',
'yref': 'y'},
{'font': {'color': '#000000'},
'showarrow': False,
'text': '0',
'x': 'rs7192',
'xref': 'x',
'y': 'rs7192',
'yref': 'y'}],
'template': '...',
'title': {'text': ('\ndefines color: r_square ░\nLD ' ... 'le, female ░\npopulations: EUR\n')},
'xaxis': {'dtick': 1,
'gridcolor': 'rgb(0, 0, 0)',
'side': 'bottom',
'ticks': '',
'title': {'font': {'size': 10},
'text': ('\nсделано с помощью ld_triangle' ... '6">поддержать разработчика</a>')}},
'yaxis': {'autorange': 'reversed', 'dtick': 1, 'ticks': '', 'ticksuffix': ' '}}
})
Наиболее значимые компоненты словареподобного объекта Figure - data и layout. Первый посвящён закладывающим основу диаграммы данным и тесно с ними связанным настройкам, второй позволяет колдовать над дизайном диаграммы. ld_heatmap['data'][0]['z'] - собственно, посчитанные для всех пар вариантов значения LD. ['data'][0]['x'] и ['data'][0]['y'] - надписи к осям, а именно, rsIDs. ['data'][0]['hovertext'] ведёт к двумерному массиву отформатированных с помощью HTML всплывающих характеристик каждой пары. ['layout']['annotations'] - вписываемые в квадратики значения LD и соответствующие настройки. ['layout']['title']['text'] - заголовок всего хитмэпа, куда помещаются биологические характеристики последнего. ['layout']['xaxis']['title']['text'] - заголовок оси X, внезапно приспособленный мною под футер с полезными ссылками. ['layout']['yaxis']['autorange'] при значении reversed переворачивает диаграмму по оси Y, что делается в нашем случае для унификации с хитмэпами LDmatrix.
В предыдущем абзаце, как видите, я ссылался на различные атрибуты чисто питоновским способом - обращением к словарным ключам и индексам элементов списков. Изменять атрибуты можно таким же макаром. Давайте, к примеру, компактизируем лейблы осей.
ld_heatmap['layout']['xaxis']['tickfont']['size'] = 10
ld_heatmap['layout']['yaxis']['tickfont']['size'] = 10
Экземпляр класса Figure разрешает нам также задействовать ООП-синтаксис, чем не могут похвастать обычные словари.
ld_heatmap.layout.xaxis.tickfont.size = 10
ld_heatmap.layout.yaxis.tickfont.size = 10
Есть ещё замечательный синтаксический сахар - функция обновления layout.
ld_heatmap.update_layout(xaxis_tickfont_size=10)
ld_heatmap.update_layout(yaxis_tickfont_size=10)
Будьте осторожны: редактирование более высокоуровневых слоёв приводит к сбросу более низкоуровневых до значений по умолчанию. Например, вот так не делайте, иначе потеряете все отличные от кегля лейблов твики осей:
ld_heatmap.layout.xaxis = {'tickfont': {'size': 10}}
ld_heatmap.layout.yaxis = {'tickfont': {'size': 10}}
Послесловие. Любые атрибуты можно точно также легко доставать и править. При внимательном разглядывании полной редакции того или иного plotly-объекта можно даже совсем забыть про статьи и форумы.