라이신
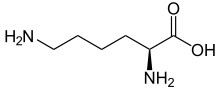 L-리신의 골격 구조식
| |||
| |||
| 이름 | |||
|---|---|---|---|
| IUPAC 이름
L-lysine
D-lysine | |||
| 체계명
(2S)-2,6-diaminohexanoic acid (L-lysine)
(2R)-2,6-diaminohexanoic acid (D-lysine) | |||
| 별칭
lysine,
D-lysine, L-lysine, LYS, h-Lys-OH | |||
| 식별자 | |||
3D 모델 (JSmol)
|
| ||
| ChEBI | |||
| ChEMBL | |||
| ChemSpider | |||
| ECHA InfoCard | 100.000.673 | ||
| KEGG | |||
PubChem CID
|
|||
| UNII |
| ||
CompTox Dashboard (EPA)
|
|||
| |||
| |||
| 성질 | |||
| C6H14N2O2 | |||
| 몰 질량 | 146.190 g·mol−1 | ||
| 1.5 kg/L | |||
| 약리학 | |||
| B05XB03 (WHO) | |||
달리 명시된 경우를 제외하면, 표준상태(25 °C [77 °F], 100 kPa)에서 물질의 정보가 제공됨.
| |||
리신(영어: lysine) (기호: Lys or K)[2]은 단백질의 생합성에 사용되는 α-아미노산이다. 리신은 α-아미노기(생물학적 조건에서 양성자화된 −NH3+ 형태), α-카복실기(생물학적 조건에서 탈양성자화된 −COO− 형태) 및 곁사슬인 리실기((CH2)4NH2)를 포함하고 있다. 리신은 염기성 양전하(생리학적 pH에서), 지방족 아미노산으로 분류된다. 리신은 AAA, AAG 코돈에 의해 암호화되어 있다. 거의 모든 다른 아미노산들과 마찬가지로 α-탄소는 카이랄성이며, 리신은 2가지 거울상 이성질체 또는 2가지를 모두 포함하는 라세미 혼합물을 나타낼 수 있다. 이 문서에서 리신은 생물학적으로 활성인 거울상 이성질체인 L-리신을 의미하며, 여기서 α-탄소는 S 입체배치이다.
인체는 리신을 합성할 수 없다. 리신은 사람에서 필수 아미노산이며, 따라서 식사를 통해 섭취해야 한다. 리신을 합성하는 생물에서는 두 가지 주요 생합성 경로인 다이아미노피멜산 경로 및 α-아미노아디프산 경로가 존재하며, 이들 경로는 별개의 효소와 기질을 사용하고 다양한 생물에서 발견된다. 리신의 이화작용은 여러 대사 경로들 중 하나를 통해 일어나며, 이 중 가장 일반적인 경로는 사카로핀 경로이다.
리신의 역할은 가장 중요하게는 단백질 생성 뿐만 아니라 콜라겐 폴리펩타이드의 가교 형성, 필수 미네랄 영양소의 흡수, 지방산 대사의 핵심적인 화합물인 카르니틴의 생성하는 역할을 한다. 리신은 또한 종종 히스톤 변형에 관여하기 때문에 후성유전체에 영향을 미친다. ε-아미노기는 종종 수소 결합에 참여하고 촉매 작용의 일반 염기로 사용된다. ε-아미노기는 카복실기와 결합하고 있는 α-탄소로부터 4번째 탄소에 부착된다.[3]
여러 생물학적 과정에서 리신의 중요성으로 인해 리신이 부족하면 결합 조직의 결함, 지방산 대사의 장애, 빈혈 및 전신 단백질 에너지 결핍을 비롯한 여러 질병 상태가 발생할 수 있다. 대조적으로 비효율적인 이화작용으로 인한 과도한 리신은 심각한 신경 질환을 유발할 수 있다.
리신은 1889년에 독일의 생화학자 페르디난트 하인리히 에드문트 드레흐셀(Ferdinand Heinrich Edmund Drechsel)에 의해 우유의 카제인 단백질에서 처음으로 분리되었다.[4] 그는 분리한 물질을 "lysin"이라고 명명했다.[5] 1902년에 독일의 화학자 에밀 피셔와 프리츠 바이게르트(Fritz Weigert)는 합성을 통해 리신의 화학 구조를 결정했다.[6]
생합성
[편집] |
리신 합성에 대한 두 가지 경로가 자연계에서 확인되었다. 다이아미노피멜산(DAP) 경로는 아스파르트산 유도 생합성 패밀리에 속하며, 트레오닌, 메티오닌 및 아이소류신의 합성에도 관여한다.[7][8] 반면 α-아미노아디프산(AAA) 경로는 글루탐산 생합성 패밀리의 일부이다.[9][10]
다이아미노피멜산 경로는 원핵생물과 식물 모두에서 발견되며 다이하이드로다이피콜린산 생성효소(DHDPS) (EC 4.3.3.7)에 의해 시작되어 아스파르트산 유래 L-아스파르트산 세미알데하이드 및 피루브산 간의 축합 반응을 통해 (4S)-4-하이드록시-2,3,4,5-테트라하이드로-(2S)-다이피콜린산(HTPA)을 생성한다.[11][12][13][14][15] 그런 다음 생성물은 전자 공여체인 NAD(P)H와 함께 다이하이드로다이피콜린산 환원효소(DHDPR) (EC 1.3.1.26)에 의해 환원되어 2,3,4,5-테트라하이드로다이피콜린산(THDP)을 생성한다.[16] 이 지점부터 아세틸레이스, 아미노기전이효소, 탈수소효소, 석시닐레이스 경로의 4가지 경로 변형이 발견되었다.[7][17] 아세틸레이스 및 석시닐레이스 변형 경로는 둘 다 4개의 효소 촉매 단계를 사용하고, 아미노기전이효소 경로는 2개의 효소를 사용하며, 탈수소효소 경로는 단일 효소를 사용한다.[18] 이들 4가지 변형 경로는 두 번째 생성물인 메소-다이아미노피멜산의 형성으로 수렴되며, 이는 후속적으로 다이아미노피멜산 탈카복실화효소(DAPDC) (EC 4.1.1.20)에 의해 촉매되는 비가역적 반응에서 효소적으로 탈카복실화되어 L-리신을 생성한다.[19][20] 다이아미노피멜산 경로는 다이하이드로다이피콜린산 생성효소가 촉매하는 축합 단계 뿐만 아니라 아스파르트산 처리에 관여하는 효소를 포함한 여러 단계에서 조절된다.[20][21] 리신은 이러한 효소에 강력한 음성 피드백 루프를 제공하고, 그 후 전체 경로를 조절한다.[21]
α-아미노아디프산 경로는 L-리신 합성을 위한 대사 중간생성물인 α-아미노아디프산을 통한 α-케토글루타르산과 아세틸-CoA의 축합을 포함한다. 이 대사 경로는 여러 효모 종과 원생생물 및 고등 균류에 존재하는 것으로 밝혀졌다.[10][22][23][24][25][26][27] 테르무스 테르모필루스(Thermus thermophilus)와 피로코쿠스 호리코시이(Pyrococcus horikoshii)에서 α-아미노아디프산 경로의 대체 변이 경로가 발견된 것으로 보고되었으며, 이는 이 경로가 원래 제안된 것보다 원핵생물에 더 널리 분포하는 것임을 나타낼 수 있다.[28][29][30] α-아미노아디프산 경로의 첫 번째 단계이자 속도 제한 단계는 아세틸-CoA와 α-케토글루타르산 사이의 축합 반응으로 호모시트르산 생성효소(EC 2.3.3.14)에 의해 촉매되어 대사 중간생성물인 호모시트릴-CoA를 생성하며, 호모시트릴-CoA는 동일한 효소인 호모시트르산 생성효소에 의해 가수분해되어 호모시트르산을 생성한다.[31] 호모시트르산은 호모아코니트산 수화효소(EC 4.2.1.36)에 의해 효소적으로 탈수되어 시스-호모아코니트산을 생성한다.[32] 그런 다음 호모아코니트산 수화효소는 시스-호모아코니트산을 재수화시켜 호모아이소시트르산을 생성하는 두 번째 반응을 촉매한다.[10] 생성물은 호모아이소시트르산 탈수소효소(EC 1.1.1.87)에 의한 산화적 탈카복실화를 거쳐 α-케토아디프산을 생성한다.[10] 그런 다음 α-아미노아디프산은 아미노기 공여체로 글루탐산을 사용하는 피리독살 인산 의존성 아미노기전이효소인 2-아미노아디프산 아미노기전이효소(EC 2.6.1.39)에 의해 형성된다.[31] 이 지점부터 α-아미노아디프산 경로는 계에 따라 차이가 난다. 균류에서 α-아미노아디프산은 홀로-(아실 운반 단백질) 생성효소(EC 2.7.8.7)에 의해 활성화되는 아데닐릴화 및 환원을 모두 포함하는 독특한 과정에서 L-2-아미노아디프산 환원효소(EC 1.2.1.95)를 통해 α-아미노아디프산-세미알데하이드로 환원된다.[10] 세미알데하이드가 형성되면 사카로핀 탈수소효소 (NADP+, L-글루탐산 형성) (EC 1.5.1.10)는 글루탐산 및 전자 공여체인 NAD(P)H와의 축합 반응을 촉매하고 이민은 환원되어 끝에서 두 번째 생성물인 사카로핀을 생성한다.[30] 균류에서 대사 경로의 마지막 단계는 사카로핀 탈수소효소 (NADP+, L-리신 형성) (EC 1.5.1.8)에 의한 사카로핀의 산화적 탈아미노화를 포함하며 L-리신을 생성한다.[10] 일부 원핵생물에서 발견되는 변형 α-아미노아디프산 경로에서 α-아미노아디프산은 먼저 N-아세틸-α-아미노아디프산으로 전환되고, 인산화된 다음 환원적으로 탈인산화되어 ε-알데하이드로 전환된다.[30][31] 그런 다음 알데하이드는 N-아세틸-리신으로 아미노기 전이되고, 이는 탈아세틸화되어 L-리신을 생성한다.[30][31] 그러나 이러한 변이 경로에 관여하는 효소는 추가적인 검증이 필요하다.
분해
[편집]
다른 모든 아미노산들과 마찬가지로 리신의 분해는 식이 리신의 섭취 또는 세포 내 단백질의 분해로부터 시작된다. 리신의 분해는 또한 유리 리신의 세포 내 농도를 조절하고 과도한 유리 리신의 독성 효과를 방지하기 위해 정상 상태를 유지하는 수단으로 사용된다.[33] 리신의 분해와 관련된 여러 경로가 있지만 가장 일반적으로 사용되는 경로는 사카로핀 경로이며, 주로 동물의 간(및 동등한 기관), 특히 미토콘드리아 내에서 일어난다.[34][33][35][36] 이 대사 경로는 앞에서 설명한 α-아미노아디프산 경로의 반대 과정이다.[34][37] 동물과 식물에서 사카로핀 경로의 처음 두 단계는 리신-케토글루타르산 환원효소와 사카로핀 탈수소효소 (NADP+, L-리신 형성) (EC 1.5.1.8) 활성을 둘 다 가지고 있는 이기능성 효소인 α-아미노아디프산 세미알데하이드 생성효소에 의해 촉매된다. 반면에 세균과 균류와 같은 다른 생물에서는 이들 두 효소가 모두 별도의 유전자에 의해 암호화된다.[38][39] 첫 번째 단계는 NAD(P)H가 전자 공여체로 작용하여 사카로핀을 생성하기 위해 α-케토글루타르산의 존재 하에서 리신-케토글루타르산 환원효소에 의한 L-리신의 환원을 포함한다.[40] 그런 다음 사카로핀은 NAD+의 존재 하에서 사카로핀 탈수소효소 (NADP+, L-리신 형성)에 의해 촉매되는 탈수 반응을 거쳐 α-아미노아디프산 세미알데하이드와 글루탐산을 생성한다.[41] L-아미노아디프산-세미알데하이드 탈수소효소 (EC 1.2.1.31)는 분자를 α-아미노아디프산으로 추가적으로 탈수시킨다.[40] 결과적으로 2-아미노아디프산 아미노기전이효소는 α-아미노아디프산 생합성 경로의 역반응을 촉매하여 α-아미노아디프산이 α-케토아디프산으로 전환되도록 한다. 생성물인 α-케토아디프산은 NAD+ 및 조효소 A의 존재 하에 탈카복실화되어 글루타릴-CoA를 생성하지만, 이와 관련된 효소는 아직 완전히 밝혀지지 않았다.[42][43] 일부 증거는 α-케토글루타르산 탈수소효소 복합체 (EC 1.2.4.2)의 E1 소단위체와 구조적으로 상동인 2-옥소아디프산 탈수소효소 복합체가 탈탄산 반응을 담당한다는 것을 시사한다.[42][44] 마지막으로 글루타릴-CoA는 글루타릴-CoA 탈수소효소 (EC 1.3.8.6)에 의해 크로토닐-CoA로 산화적 탈카복실화되며, 이는 시트르산 회로에 관여하는 필수 탄소 대사 산물인 아세틸-CoA를 생성하기 위해 여러 효소 단계를 거쳐 추가적으로 처리된다.[40][45][46][47]
영양가
[편집]리신은 사람에서 필수 아미노산이다.[48] 사람의 영양 요구량은 유아기의 약 60 mg/kg에서 30 mg/kg까지 다양하다.[34] 이러한 요구량은 일반적으로 권장 요구량을 훨씬 초과하는 육류 및 채소 공급원으로부터 리신을 섭취하는 서구 사회에서 충족된다.[34] 채식주의 식단에서 리신의 섭취량은 육류에 비해 곡물의 제한된 리신의 양으로 인해 더 적다.[34]
곡물의 리신의 양이 제하되어 있다는 것을 감안하여 유전자 변형을 통해 리신의 함량이 증가될 수 있을 것이라고 오랫동안 추측되어 왔다.[49][50] 종종 이러한 유전자 변형은 다이하이드로다이피콜린산 생성효소의 리신 피드백-민감성 오르토로그를 도입함으로써 다이아미노피멜산 경로의 의도적인 조절 장애를 수반한다.[49][50] 이러한 방법은 증가된 유리 리신의 독성 부작용과 시트르산 회로에 대한 간접적인 영향으로 인해 제한된 성공을 거두었다.[51] 식물은 식물 종자 내에서 발견되는 저장 단백질의 형태로 리신 및 기타 아미노산을 축적하며, 이는 작물의 식용 성분을 나타낸다.[52] 이것은 유리 리신을 증가시킬 뿐만 아니라 안정한 종자 단백질의 합성을 위해 리신을 직접적으로 증가시킬 필요가 있음을 강조하고, 결과적으로 작물의 소비성 성분의 영양 가치를 증가시킨다.[53][54] 유전자 변형은 제한적인 성공을 거두었지만, 보다 전통적인 선택적 육종 기술은 필수 아미노산인 리신과 트립토판의 수준을 크게 증가시킨 품질 단백질 옥수수의 분리를 가능하게 했다. 이러한 리신의 함량 증가는 리신 결핍 제인 관련 종자 저장 단백질의 전사를 감소시키고 결과적으로 리신이 풍부한 다른 단백질의 함량을 증가시키는 불투명-2 돌연변이에 기인한다.[54][55] 일반적으로 가축 사료에 제한적으로 존재하는 리신을 보충하기 위해 산업적으로 생산된 리신을 첨가한다.[56][57] 산업적 공정에는 코리네박테리움 글루타미쿰(Corynebacterium glutamicum)의 발효 배양과 리신의 후속 정제가 포함된다.[56]
식이 공급원
[편집]리신의 좋은 공급원으로는 계란, 육류(특히 붉은 고기, 양고기, 돼지고기, 가금류), 콩, 완두, 치즈(특히 파르미자노 레자노), 특정 생선(대구 및 정어리 등)과 같은 고단백 식품들이 있다.[58] 리신은 대부분의 곡물에서 제한 아미노산(특정 식품에서 가장 적은 양으로 발견되는 필수 아미노산)이지만, 대부분의 콩류에는 풍부하다.[59] 콩에는 옥수수에 없는 리신이 포함되어 있으며, 고고학 기록에서 콩, 옥수수, 호박의 세 자매처럼 콩과 옥수수가 함께 등장하는 경우가 많다.[60]
단백질 1 g당 최소 51 mg의 리신을 포함하고 있다면(단백질이 5.1%의 리신을 포함하도록) 식품에 충분한 양의 리신이 있는 것으로 간주된다.[61] L-리신 HCl은 80.03%의 L-리신을 제공하는 식이 보충제로 사용된다.[62] 이와 같이 L-리신 HCl 1.25 g에는 L-리신 1 g이 함유되어 있다.
생물학적 역할
[편집]리신의 가장 일반적인 역할을 단백질 생성에 참여하는 것이다. 리신은 종종 단백질의 구조에서 중요한 역할을 한다. 리신의 곁사슬은 한쪽 끝에 양전하를 띤 작용기와 골격에 가까운 긴 소수성 탄소 꼬리를 포함하고 있기 때문에 리신은 다소 양친매성으로 간주된다. 이러한 이유로 리신은 수용성 환경과 상호작용할 수 있는 용매 채널 및 단백질 외부에서도 더 일반적으로 묻혀 있는 상태로 발견된다.[63] 리신은 또한 ε-아미노기가 종종 수소 결합, 염다리 및 공유결합성 상호작용에 참여하여 시프 염기를 형성하기 때문에 단백질의 안정성에 기여할 수 있다.[63][64][65][66]
리신의 두 번째 주요 역할은 히스톤 변형을 통한 후성유전적 조절이다.[67][68] 히스톤의 돌출된 꼬리에서 발견되는 리신 잔기를 일반적으로 포함하는 공유결합성 히스톤 변형에는 여러 유형이 존재한다. 이러한 변형에는 종종 아세틸기(−CH3CO)를 첨가 또는 제거하여 아세틸리신을 형성하거나 리신으로 되돌리는 것, 최대 3개의 메틸기(−CH3), 유비퀴틴 또는 SUMO 단백질을 첨가 또는 제거하는 것이 포함된다.[67][69][70][71][72] 다양한 변형은 유전자가 활성화되거나 억제될 수 있는 유전자 발현의 조절에서 하류 효과를 가지게 할 수 있다.
리신은 또한 결합 조직의 구조 단백질, 칼슘의 항상성, 지방산 대사를 포함한 다른 생물학적 과정에서 중요한 역할을 하는 것으로 알려져 있다.[73][74][75] 리신은 콜라겐에 있는 3개의 나선형 폴리펩타이드 사이의 가교 형성에 관여하여 안정성과 인장 강도에 기여하는 것으로 나타났다.[73][76] 이러한 메커니즘은 세균의 세포벽에서 리신의 역할과 유사하며, 여기서 리신(및 메소-다이아미노피멜산)은 가교 형성 및 세포벽의 안정성 유지에 중요하다.[77] 이 개념은 이전에 잠재적으로 병원성인 유전자 변형 세균의 원치 않는 방출을 피하기 위한 수단으로 연구되었다. 대장균의 영양요구성 균주(X1776)는 다이아미노피멜산의 보충없이는 생존할 수 없고 따라서 실험실 환경 밖에서는 살 수 없기 때문에 모든 유전자 변형 실험에 사용될 수 있다고 제안되었다.[78] 리신은 또한 장에서 칼슘 흡수 및 콩팥에서의 유지에 관여하는 것으로 제안되었으며, 따라서 칼슘의 항상성 유지에 역할을 할 수 있다.[74] 마지막으로 리신은 지방산을 산화시켜 에너지를 생성하기 위해 지방산을 미토콘드리아로 운반하는 카르니틴의 전구체인 것으로 나타났다.[75][79] 카르니틴은 특정 단백질의 분해 산물인 트라이메틸리신으로부터 합성된다. 이러한 리신은 먼저 단백질로 통합되고 카르니틴으로 전환되기 전에 메틸화되어야 한다.[75] 그러나 포유류에서 카르니틴의 주요 공급원은 리신의 전환에 의한 것보다는 식이 공급원의 섭취에 의한 것이다.[75]
로돕신과 같은 옵신 및 시각 옵신(OPN1SW, OPN1MW 및 OPN1LW 유전자에 의해 암호화 됨)에서 레티날은 보존된 리신 잔기와 함께 시프 염기를 형성하고 빛과 레티닐리덴기의 상호작용은 색각에서 신호 변환을 유발한다.
논쟁의 여지가 있는 역할
[편집]리신이 정맥 내 또는 경구 투여될 때 성장 호르몬의 방출을 상당히 증가시킬 수 있다는 논의가 오랫동안 있어 왔다.[80] 이러한 논의 덕분에 운동 선수들이 훈련 중에 근육 성장을 촉진하는 수단으로 리신을 사용하게 되었지만, 리신의 이러한 작용을 뒷받침하는 중요한 증거는 현재까지 발견되지 않았다.[80][81]
단순포진 바이러스(HSV) 단백질은 이들이 감염시키는 세포의 단백질보다 아르기닌이 풍부하고 리신이 부족하기 때문에 리신 보충제가 치료제로 사용할 수 있는지 여부가 시도되었다. 이들 두 가지 아미노산은 장에서 흡수되고 콩팥에서 재흡수되며 동일한 아미노산 수송체에 의해 세포로 이동되기 때문에 이론상 풍부한 리신은 바이러스 복제에 사용할 수 있는 아르기닌의 양을 제한한다.[82] 임상 연구에 따르면 리신의 보충이 단순포진 바이러스에 의한 발병에 대한 예방 또는 치료에 대해 효과가 있다는 좋은 증거는 발견되지 않았다.[83][84] 리신이 단순포진 바이러스에 대한 면역 반응을 개선할 수 있다는 주장에 대해 유럽 식품안전청의 리뷰 결과에서 인과 관계에 의한 증거는 발견되지 않았다. 2011년에 발표된 동일한 리뷰에서는 리신이 콜레스테롤을 낮추고, 식욕을 증가시키며, 일반 영양소 이외의 다른 역할에서 단백질 합성에 기여하거나, 칼슘 흡수 또는 보유를 증가시킬 수 있다는 주장을 뒷받침하는 증거를 발견하지 못했다.[85]
질병에서의 역할
[편집]리신과 관련된 질병은 리신의 하류 처리, 즉 단백질로의 통합 또는 대체 생체분자로의 변형의 결과이다. 콜라겐에서 리신의 역할은 위에서 설명했듯이 콜라겐 펩타이드의 가교 형성에 관여하는 리신과 하이드록시리신의 결핍은 결합 조직의 질병 상태와 관련이 있다.[86] 카르니틴은 지방산 대사에 관여하는 주요 리신 유래 대사 산물이기 때문에 충분한 카르니틴과 리신이 부족한 표준 이하의 식단은 카르니틴의 수치를 감소시켜 개인의 건강에 상당한 연쇄 영향을 미칠 수 있다.[79][87] 리신은 철분의 흡수에 영향을 미치는 것으로 의심되고, 그 결과 혈장 내 페리틴의 농도에 영향을 미치는 것으로 의심되기 때문에 리신은 빈혈에도 중요한 역할을 하는 것으로 나타났다.[88] 그러나 정확한 작용 메커니즘은 아직 밝혀지지 않았다.[88] 가장 일반적으로 리신의 결핍은 비서구 사회에서 찾아볼 수 있으며, 개인의 건강에 중대하고 전신적인 영향을 미치는 단백질-에너지 영양실조로 나타난다.[89][90] 리신의 분해를 담당하는 효소, 즉 사카로핀 경로의 이기능성 α-아미노아디프산 세미알데하이드 생성효소의 돌연변이와 관련된 유전 질환도 있다.[91] 리신 이화작용의 결핍으로 인해 아미노산이 혈장에 축적되고 환자에게서 뇌전증, 운동실조, 경직 및 정신운동 지체를 포함한 심각한 신경질환에 무증상으로 나타날 수 있는 고리신혈증이 발병할 수 있다.[91][92] 고리신혈증의 임상적 중요성은 신체적 또는 정신적 장애와 고리신혈증 사이에 상관관계가 없다는 일부 연구로 인해 현장에서 논쟁의 대상이다.[93] 이 외에도 리신 대사와 관련된 유전자의 돌연변이는 피리독신 의존성 뇌전증(ALDH7A1 유전자), α-아미노아디프산뇨증 및 α-케토아디프산뇨증(DHTKD1 유전자), 글루타르산뇨증 1형(GCDH 유전자)을 비롯한 여러 질병 상태와 관련이 있다.[42][94][95][96][97]
고리신혈증은 소변에 다량의 리신이 발견된다.[98] 고리신혈증은 리신의 분해에 관여하는 단백질이 유전적 돌연변이로 인해 기능을 하지 못하는 대사성 질환으로 인해 발병하는 경우가 많다.[99] 고리신혈증은 또한 세뇨관 수송의 실패로 인해 발생할 수 있다.[99]
동물 사료에 리신의 사용
[편집]동물 사료용 리신의 생산은 2009년에 거의 700,000톤에 달하는 주요 글로벌 산업이며, 시장 가치는 12억 2천만 유로(€) 이상이다.[100] 리신은 육류 샌산을 위해 돼지 및 닭과 같은 특정 동물의 생장을 최적화할 때의 제한 아미노산이기 때문에 동물 사료에 중요한 첨가제이다. 리신을 보충하면 높은 생장률을 유지하고 질소 배설로 인한 오염을 제한하면서 저렴한 식물성 단백질(예: 콩보다 옥수수)을 사용할 수 있다.[101] 그러나 옥수수가 가금류 및 돼지 사료로 사용될 때 주요 환경 비용은 인산염에 의한 오염이다.[102]
리신은 주로 당류를 기반으로 미생물 발효에 의해 산업적으로 생산된다. 유전공학에서는 생산 효율을 높이고 다른 기질로부터 리신을 합성할 수 있는 세균 균주를 적극적으로 얻으려고 애쓰고 있다.[100]
대중문화에서
[편집]1993년의 영화 《쥬라기 공원》(1990년의 마이클 크라이튼의 동명의 소설을 원작으로 함)에서는 조작된 영양요구변이체의 한 예로 리신을 생산할 수 없도록 유전적으로 변형된 공룡들이 묘사된다.[103] 이것은 "리신 우발상황(lysine contingency)"으로 알려졌으며 복제된 공룡들이 공원 밖에서 살아남는 것을 방지하기 위해 공원의 수의사가 제공하는 리신 보충제에 의존하도록 했다. 실제로 리신(필수 아미노산)을 생산할 수 있는 동물은 없다.[104]
1996년에 리신은 미국 역사상 가장 큰 가격 담합 사건에서 초점이 되었다. 아처 대니얼스 미들랜드 컴퍼니는 1억 달러의 벌금을 냈고, 경영진 중 3명은 유죄 판결을 받고 복역했다. 또한 가격 담합 사건에서 일본 기업 2곳(아지노모토, 교와 핫코)과 한국 기업(세원)이 유죄 판결을 받았다.[105] 리신의 가격을 담합한 공모자들의 비밀 비디오 녹화물은 온라인이나 미국 법무부 독점금지과에 비디오를 요청하여 찾아볼 수 있다. 이 사건은 영화 《인포먼트》와 동명의 책의 바탕이 되었다.[106]
같이 보기
[편집]각주
[편집]- ↑ 가 나 Williams, P. A.; Hughes, C. E.; Harris, K. D. M (2015). “L‐Lysine: Exploiting Powder X‐ray Diffraction to Complete the Set of Crystal Structures of the 20 Directly Encoded Proteinogenic Amino Acids”. 《Angew. Chem. Int. Ed.》 54 (13): 3973–3977. doi:10.1002/anie.201411520. PMID 25651303.
- ↑ “IUPAC-IUB Joint Commission on Biochemical Nomenclature (JCBN). Nomenclature and symbolism for amino acids and peptides. Recommendations 1983”. 《Biochemical Journal》 219 (2): 345–373. 1984년 4월 15일. doi:10.1042/bj2190345. PMC 1153490. PMID 6743224.
- ↑ Lysine. The Biology Project, Department of Biochemistry and Molecular Biophysics, University of Arizona.
- ↑ Drechsel E (1889). “Zur Kenntniss der Spaltungsprodukte des Caseïns” [[Contribution] to [our] knowledge of the cleavage products of casein]. 《Journal für Praktische Chemie》. 2nd series (독일어) 39: 425–429. doi:10.1002/prac.18890390135. On p. 428, Drechsel presented an empirical formula for the chloroplatinate salt of lysine – C8H16N2O2Cl2·PtCl4 + H2O – but he later admitted that this formula was wrong because the salt's crystals contained ethanol instead of water. See: Drechsel E (1891). “Der Abbau der Eiweissstoffe” [The disassembly of proteins]. 《Archiv für Anatomie und Physiologie》 (독일어): 248–278.; Drechsel E (1877). “Zur Kenntniss der Spaltungsproducte des Caseïns” [Contribution] to [our] knowledge of the cleavage products of casein] (독일어): 254–260.
From p. 256:] " … die darin enthaltene Base hat die Formel C6H14N2O2. Der anfängliche Irrthum ist dadurch veranlasst worden, dass das Chloroplatinat nicht, wie angenommen ward, Krystallwasser, sondern Krystallalkohol enthält, … " ( … the base [that's] contained therein has the [empirical] formula C6H14N2O2. The initial error was caused by the chloroplatinate containing not water in the crystal (as was assumed), but ethanol … )
- ↑ Drechsel E (1891). “Der Abbau der Eiweissstoffe” [The disassembly of proteins]. 《Archiv für Anatomie und Physiologie》 (독일어): 248–278.; Fischer, Ernst (1891). “Ueber neue Spaltungsproducte des Leimes” [On new cleavage products of gelatin] (독일어): 465–469.
From p. 469:] " … die Base C6H14N2O2, welche mit dem Namen Lysin bezeichnet werden mag, … " ( … the base C6H14N2O2, which may be designated with the name "lysine", … ) [Note: Ernst Fischer was a graduate student of Drechsel.]
- ↑ Fischer, Emil; Weigert, Fritz (1902). “Synthese der α,ε – Diaminocapronsäure (Inactives Lysin)” [Synthesis of α,ε-diaminohexanoic acid ([optically] inactive lysine)]. 《Berichte der Deutschen Chemischen Gesellschaft》 (독일어) 35 (3): 3772–3778. doi:10.1002/cber.190203503211.
- ↑ 가 나 Hudson AO, Bless C, Macedo P, Chatterjee SP, Singh BK, Gilvarg C, Leustek T (January 2005). “Biosynthesis of lysine in plants: evidence for a variant of the known bacterial pathways”. 《Biochimica et Biophysica Acta (BBA) - General Subjects》 1721 (1–3): 27–36. doi:10.1016/j.bbagen.2004.09.008. PMID 15652176.
- ↑ Velasco AM, Leguina JI, Lazcano A (October 2002). “Molecular evolution of the lysine biosynthetic pathways”. 《Journal of Molecular Evolution》 55 (4): 445–459. Bibcode:2002JMolE..55..445V. doi:10.1007/s00239-002-2340-2. PMID 12355264. S2CID 19460256.
- ↑ Miyazaki T, Miyazaki J, Yamane H, Nishiyama M (July 2004). “alpha-Aminoadipate aminotransferase from an extremely thermophilic bacterium, Thermus thermophilus” (PDF). 《Microbiology》 150 (Pt 7): 2327–2334. doi:10.1099/mic.0.27037-0. PMID 15256574. S2CID 25416966. 2020년 2월 28일에 원본 문서 (PDF)에서 보존된 문서.
- ↑ 가 나 다 라 마 바 Xu H, Andi B, Qian J, West AH, Cook PF (2006). “The alpha-aminoadipate pathway for lysine biosynthesis in fungi”. 《Cell Biochemistry and Biophysics》 46 (1): 43–64. doi:10.1385/CBB:46:1:43. PMID 16943623. S2CID 22370361.
- ↑ Atkinson SC, Dogovski C, Downton MT, Czabotar PE, Dobson RC, Gerrard JA, Wagner J, Perugini MA (March 2013). “Structural, kinetic and computational investigation of Vitis vinifera DHDPS reveals new insight into the mechanism of lysine-mediated allosteric inhibition”. 《Plant Molecular Biology》 81 (4–5): 431–446. doi:10.1007/s11103-013-0014-7. hdl:11343/282680. PMID 23354837. S2CID 17129774.
- ↑ Griffin MD, Billakanti JM, Wason A, Keller S, Mertens HD, Atkinson SC, Dobson RC, Perugini MA, Gerrard JA, Pearce FG (2012). “Characterisation of the first enzymes committed to lysine biosynthesis in Arabidopsis thaliana”. 《PLOS ONE》 7 (7): e40318. Bibcode:2012PLoSO...740318G. doi:10.1371/journal.pone.0040318. PMC 3390394. PMID 22792278.
- ↑ Soares da Costa TP, Muscroft-Taylor AC, Dobson RC, Devenish SR, Jameson GB, Gerrard JA (July 2010). “How essential is the 'essential' active-site lysine in dihydrodipicolinate synthase?”. 《Biochimie》 92 (7): 837–845. doi:10.1016/j.biochi.2010.03.004. PMID 20353808.
- ↑ Soares da Costa TP, Christensen JB, Desbois S, Gordon SE, Gupta R, Hogan CJ, Nelson TG, Downton MT, Gardhi CK, Abbott BM, Wagner J, Panjikar S, Perugini MA (2015). 〈Quaternary Structure Analyses of an Essential Oligomeric Enzyme〉. 《Analytical Ultracentrifugation》. Methods in Enzymology 562. 205–223쪽. doi:10.1016/bs.mie.2015.06.020. ISBN 9780128029084. PMID 26412653.
- ↑ Muscroft-Taylor AC, Soares da Costa TP, Gerrard JA (March 2010). “New insights into the mechanism of dihydrodipicolinate synthase using isothermal titration calorimetry”. 《Biochimie》 92 (3): 254–262. doi:10.1016/j.biochi.2009.12.004. PMID 20025926.
- ↑ Christensen JB, Soares da Costa TP, Faou P, Pearce FG, Panjikar S, Perugini MA (November 2016). “Structure and Function of Cyanobacterial DHDPS and DHDPR”. 《Scientific Reports》 6 (1): 37111. Bibcode:2016NatSR...637111C. doi:10.1038/srep37111. PMC 5109050. PMID 27845445.
- ↑ McCoy AJ, Adams NE, Hudson AO, Gilvarg C, Leustek T, Maurelli AT (November 2006). “L,L-diaminopimelate aminotransferase, a trans-kingdom enzyme shared by Chlamydia and plants for synthesis of diaminopimelate/lysine”. 《Proceedings of the National Academy of Sciences of the United States of America》 103 (47): 17909–17914. Bibcode:2006PNAS..10317909M. doi:10.1073/pnas.0608643103. PMC 1693846. PMID 17093042.
- ↑ Hudson AO, Gilvarg C, Leustek T (May 2008). “Biochemical and phylogenetic characterization of a novel diaminopimelate biosynthesis pathway in prokaryotes identifies a diverged form of LL-diaminopimelate aminotransferase”. 《Journal of Bacteriology》 190 (9): 3256–3263. doi:10.1128/jb.01381-07. PMC 2347407. PMID 18310350.
- ↑ Peverelli MG, Perugini MA (August 2015). “An optimized coupled assay for quantifying diaminopimelate decarboxylase activity”. 《Biochimie》 115: 78–85. doi:10.1016/j.biochi.2015.05.004. PMID 25986217.
- ↑ 가 나 Soares da Costa TP, Desbois S, Dogovski C, Gorman MA, Ketaren NE, Paxman JJ, Siddiqui T, Zammit LM, Abbott BM, Robins-Browne RM, Parker MW, Jameson GB, Hall NE, Panjikar S, Perugini MA (August 2016). “Structural Determinants Defining the Allosteric Inhibition of an Essential Antibiotic Target”. 《Structure》 24 (8): 1282–1291. doi:10.1016/j.str.2016.05.019. PMID 27427481.
- ↑ 가 나 Jander G, Joshi V (2009년 1월 1일). “Aspartate-Derived Amino Acid Biosynthesis in Arabidopsis thaliana”. 《The Arabidopsis Book》 7: e0121. doi:10.1199/tab.0121. PMC 3243338. PMID 22303247.
- ↑ Andi B, West AH, Cook PF (September 2004). “Kinetic mechanism of histidine-tagged homocitrate synthase from Saccharomyces cerevisiae”. 《Biochemistry》 43 (37): 11790–11795. doi:10.1021/bi048766p. PMID 15362863.
- ↑ Bhattacharjee JK (1985). “alpha-Aminoadipate pathway for the biosynthesis of lysine in lower eukaryotes”. 《Critical Reviews in Microbiology》 12 (2): 131–151. doi:10.3109/10408418509104427. PMID 3928261.
- ↑ Bhattacharjee JK, Strassman M (May 1967). “Accumulation of tricarboxylic acids related to lysine biosynthesis in a yeast mutant”. 《The Journal of Biological Chemistry》 242 (10): 2542–2546. doi:10.1016/S0021-9258(18)95997-1. PMID 6026248.
- ↑ Gaillardin CM, Ribet AM, Heslot H (November 1982). “Wild-type and mutant forms of homoisocitric dehydrogenase in the yeast Saccharomycopsis lipolytica”. 《European Journal of Biochemistry》 128 (2–3): 489–494. doi:10.1111/j.1432-1033.1982.tb06991.x. PMID 6759120.
- ↑ Jaklitsch WM, Kubicek CP (July 1990). “Homocitrate synthase from Penicillium chrysogenum. Localization, purification of the cytosolic isoenzyme, and sensitivity to lysine”. 《The Biochemical Journal》 269 (1): 247–253. doi:10.1042/bj2690247. PMC 1131560. PMID 2115771.
- ↑ Ye ZH, Bhattacharjee JK (December 1988). “Lysine biosynthesis pathway and biochemical blocks of lysine auxotrophs of Schizosaccharomyces pombe”. 《Journal of Bacteriology》 170 (12): 5968–5970. doi:10.1128/jb.170.12.5968-5970.1988. PMC 211717. PMID 3142867.
- ↑ Kobashi N, Nishiyama M, Tanokura M (March 1999). “Aspartate kinase-independent lysine synthesis in an extremely thermophilic bacterium, Thermus thermophilus: lysine is synthesized via alpha-aminoadipic acid not via diaminopimelic acid”. 《Journal of Bacteriology》 181 (6): 1713–1718. doi:10.1128/JB.181.6.1713-1718.1999. PMC 93567. PMID 10074061.
- ↑ Kosuge T, Hoshino T (1999). “The alpha-aminoadipate pathway for lysine biosynthesis is widely distributed among Thermus strains”. 《Journal of Bioscience and Bioengineering》 88 (6): 672–675. doi:10.1016/S1389-1723(00)87099-1. PMID 16232683.
- ↑ 가 나 다 라 Nishida H, Nishiyama M, Kobashi N, Kosuge T, Hoshino T, Yamane H (December 1999). “A prokaryotic gene cluster involved in synthesis of lysine through the amino adipate pathway: a key to the evolution of amino acid biosynthesis”. 《Genome Research》 9 (12): 1175–1183. doi:10.1101/gr.9.12.1175. PMID 10613839.
- ↑ 가 나 다 라 Nishida H, Nishiyama M (September 2000). “What is characteristic of fungal lysine synthesis through the alpha-aminoadipate pathway?”. 《Journal of Molecular Evolution》 51 (3): 299–302. Bibcode:2000JMolE..51..299N. doi:10.1007/s002390010091. PMID 11029074. S2CID 1265909.
- ↑ Zabriskie TM, Jackson MD (February 2000). “Lysine biosynthesis and metabolism in fungi”. 《Natural Product Reports》 17 (1): 85–97. doi:10.1039/a801345d. PMID 10714900.
- ↑ 가 나 Zhu X, Galili G (May 2004). “Lysine metabolism is concurrently regulated by synthesis and catabolism in both reproductive and vegetative tissues”. 《Plant Physiology》 135 (1): 129–136. doi:10.1104/pp.103.037168. PMC 429340. PMID 15122025.
- ↑ 가 나 다 라 마 Tomé D, Bos C (June 2007). “Lysine requirement through the human life cycle”. 《The Journal of Nutrition》 137 (6 Suppl 2): 1642S–1645S. doi:10.1093/jn/137.6.1642S. PMID 17513440.
- ↑ Blemings KP, Crenshaw TD, Swick RW, Benevenga NJ (August 1994). “Lysine-alpha-ketoglutarate reductase and saccharopine dehydrogenase are located only in the mitochondrial matrix in rat liver”. 《The Journal of Nutrition》 124 (8): 1215–1221. doi:10.1093/jn/124.8.1215. PMID 8064371.
- ↑ Galili G, Tang G, Zhu X, Gakiere B (June 2001). “Lysine catabolism: a stress and development super-regulated metabolic pathway”. 《Current Opinion in Plant Biology》 4 (3): 261–266. doi:10.1016/s1369-5266(00)00170-9. PMID 11312138.
- ↑ Arruda P, Kemper EL, Papes F, Leite A (August 2000). “Regulation of lysine catabolism in higher plants”. 《Trends in Plant Science》 5 (8): 324–330. doi:10.1016/s1360-1385(00)01688-5. PMID 10908876.
- ↑ Sacksteder KA, Biery BJ, Morrell JC, Goodman BK, Geisbrecht BV, Cox RP, Gould SJ, Geraghty MT (June 2000). “Identification of the alpha-aminoadipic semialdehyde synthase gene, which is defective in familial hyperlysinemia”. 《American Journal of Human Genetics》 66 (6): 1736–1743. doi:10.1086/302919. PMC 1378037. PMID 10775527.
- ↑ Zhu X, Tang G, Galili G (December 2002). “The activity of the Arabidopsis bifunctional lysine-ketoglutarate reductase/saccharopine dehydrogenase enzyme of lysine catabolism is regulated by functional interaction between its two enzyme domains”. 《The Journal of Biological Chemistry》 277 (51): 49655–49661. doi:10.1074/jbc.m205466200. PMID 12393892.
- ↑ 가 나 다 Kiyota E, Pena IA, Arruda P (November 2015). “The saccharopine pathway in seed development and stress response of maize”. 《Plant, Cell & Environment》 38 (11): 2450–2461. doi:10.1111/pce.12563. PMID 25929294.
- ↑ Serrano GC, Rezende e Silva Figueira T, Kiyota E, Zanata N, Arruda P (March 2012). “Lysine degradation through the saccharopine pathway in bacteria: LKR and SDH in bacteria and its relationship to the plant and animal enzymes”. 《FEBS Letters》 586 (6): 905–911. doi:10.1016/j.febslet.2012.02.023. PMID 22449979. S2CID 32385212.
- ↑ 가 나 다 Danhauser K, Sauer SW, Haack TB, Wieland T, Staufner C, Graf E, Zschocke J, Strom TM, Traub T, Okun JG, Meitinger T, Hoffmann GF, Prokisch H, Kölker S (December 2012). “DHTKD1 mutations cause 2-aminoadipic and 2-oxoadipic aciduria”. 《American Journal of Human Genetics》 91 (6): 1082–1087. doi:10.1016/j.ajhg.2012.10.006. PMC 3516599. PMID 23141293.
- ↑ Sauer SW, Opp S, Hoffmann GF, Koeller DM, Okun JG, Kölker S (January 2011). “Therapeutic modulation of cerebral L-lysine metabolism in a mouse model for glutaric aciduria type I”. 《Brain》 134 (Pt 1): 157–170. doi:10.1093/brain/awq269. PMID 20923787.
- ↑ Goncalves RL, Bunik VI, Brand MD (February 2016). “Production of superoxide/hydrogen peroxide by the mitochondrial 2-oxoadipate dehydrogenase complex”. 《Free Radical Biology & Medicine》 91: 247–255. doi:10.1016/j.freeradbiomed.2015.12.020. PMID 26708453.
- ↑ Goh DL, Patel A, Thomas GH, Salomons GS, Schor DS, Jakobs C, Geraghty MT (July 2002). “Characterization of the human gene encoding alpha-aminoadipate aminotransferase (AADAT)”. 《Molecular Genetics and Metabolism》 76 (3): 172–180. doi:10.1016/s1096-7192(02)00037-9. PMID 12126930.
- ↑ Härtel U, Eckel E, Koch J, Fuchs G, Linder D, Buckel W (1993년 2월 1일). “Purification of glutaryl-CoA dehydrogenase from Pseudomonas sp., an enzyme involved in the anaerobic degradation of benzoate”. 《Archives of Microbiology》 159 (2): 174–181. doi:10.1007/bf00250279. PMID 8439237. S2CID 2262592.
- ↑ Sauer SW (October 2007). “Biochemistry and bioenergetics of glutaryl-CoA dehydrogenase deficiency”. 《Journal of Inherited Metabolic Disease》 30 (5): 673–680. doi:10.1007/s10545-007-0678-8. PMID 17879145. S2CID 20609879.
- ↑ Nelson, David L; Cox, Michael M; Lehninger, Albert L (2013). 《Lehninger principles of biochemistry》 6판. New York: W.H. Freeman and Company. ISBN 978-1-4641-0962-1. OCLC 824794893.
- ↑ 가 나 Galili G, Amir R (February 2013). “Fortifying plants with the essential amino acids lysine and methionine to improve nutritional quality”. 《Plant Biotechnology Journal》 11 (2): 211–222. doi:10.1111/pbi.12025. PMID 23279001.
- ↑ 가 나 Wang G, Xu M, Wang W, Galili G (June 2017). “Fortifying Horticultural Crops with Essential Amino Acids: A Review”. 《International Journal of Molecular Sciences》 18 (6): 1306. doi:10.3390/ijms18061306. PMC 5486127. PMID 28629176.
- ↑ Angelovici R, Fait A, Fernie AR, Galili G (January 2011). “A seed high-lysine trait is negatively associated with the TCA cycle and slows down Arabidopsis seed germination”. 《The New Phytologist》 189 (1): 148–159. doi:10.1111/j.1469-8137.2010.03478.x. PMID 20946418.
- ↑ Edelman M, Colt M (2016). “Nutrient Value of Leaf vs. Seed”. 《Frontiers in Chemistry》 4: 32. doi:10.3389/fchem.2016.00032. PMC 4954856. PMID 27493937.
- ↑ Jiang SY, Ma A, Xie L, Ramachandran S (September 2016). “Improving protein content and quality by over-expressing artificially synthetic fusion proteins with high lysine and threonine constituent in rice plants”. 《Scientific Reports》 6 (1): 34427. Bibcode:2016NatSR...634427J. doi:10.1038/srep34427. PMC 5039639. PMID 27677708.
- ↑ 가 나 Shewry, Peter R. (November 2007). “Improving the protein content and composition of cereal grain”. 《Journal of Cereal Science》 46 (3): 239–250. doi:10.1016/j.jcs.2007.06.006.
- ↑ Prasanna B, Vasal SK, Kassahun B, Singh NN (2001). “Quality protein maize”. 《Current Science》 81 (10): 1308–1319. JSTOR 24105845.
- ↑ 가 나 Kircher M, Pfefferle W (April 2001). “The fermentative production of L-lysine as an animal feed additive”. 《Chemosphere》 43 (1): 27–31. Bibcode:2001Chmsp..43...27K. doi:10.1016/s0045-6535(00)00320-9. PMID 11233822.
- ↑ Junior, Letti; Alberto, Luiz; Letti, Gilberto Vinícius Melo; Soccol, Carlos Ricardo; Junior, Letti; Alberto, Luiz; Letti, Gilberto Vinícius Melo; Soccol, Carlos Ricardo (2016). “Development of an L-Lysine Enriched Bran for Animal Nutrition via Submerged Fermentation by Corynebacterium glutamicum using Agroindustrial Substrates”. 《Brazilian Archives of Biology and Technology》 59. doi:10.1590/1678-4324-2016150519. ISSN 1516-8913.
- ↑ University of Maryland Medical Center. “Lysine”. 2009년 12월 30일에 확인함.
- ↑ Young VR, Pellett PL (1994). “Plant proteins in relation to human protein and amino acid nutrition”. 《American Journal of Clinical Nutrition》 59 (5 Suppl): 1203S–1212S. doi:10.1093/ajcn/59.5.1203s. PMID 8172124. S2CID 35271281.
- ↑ Landon, Amanda J. (2008). “The 'How' of the Three Sisters: The Origins of Agriculture in Mesoamerica and the Human Niche”. 《Nebraska Anthropologist》: 110–124. 2022년 8월 9일에 확인함.
- ↑ Institute of Medicine of the National Academies (2005). 《Dietary Reference Intakes for Macronutrients》. 589쪽. doi:10.17226/10490. ISBN 978-0-309-08525-0. 2017년 10월 29일에 확인함.
- ↑ “Dietary Supplement Database: Blend Information (DSBI)”.
L-Lysine HCl 10000820 80.03% lysine
- ↑ 가 나 Betts, Matthew J.; Russell, Robert B. (2003). Barnes, Michael R.; Gray, Ian C., 편집. 《Bioinformatics for Geneticists》. John Wiley & Sons, Ltd. 289–316쪽. doi:10.1002/0470867302.ch14. ISBN 978-0-470-86730-3.
- ↑ Blickling S, Renner C, Laber B, Pohlenz HD, Holak TA, Huber R (January 1997). “Reaction mechanism of Escherichia coli dihydrodipicolinate synthase investigated by X-ray crystallography and NMR spectroscopy”. 《Biochemistry》 36 (1): 24–33. doi:10.1021/bi962272d. PMID 8993314. S2CID 23072673.
- ↑ Kumar S, Tsai CJ, Nussinov R (March 2000). “Factors enhancing protein thermostability”. 《Protein Engineering》 13 (3): 179–91. doi:10.1093/protein/13.3.179. PMID 10775659.
- ↑ Sokalingam S, Raghunathan G, Soundrarajan N, Lee SG (2012년 7월 9일). “A study on the effect of surface lysine to arginine mutagenesis on protein stability and structure using green fluorescent protein”. 《PLOS ONE》 7 (7): e40410. Bibcode:2012PLoSO...740410S. doi:10.1371/journal.pone.0040410. PMC 3392243. PMID 22792305.
- ↑ 가 나 Dambacher S, Hahn M, Schotta G (July 2010). “Epigenetic regulation of development by histone lysine methylation”. 《Heredity》 105 (1): 24–37. doi:10.1038/hdy.2010.49. PMID 20442736.
- ↑ Martin C, Zhang Y (November 2005). “The diverse functions of histone lysine methylation”. 《Nature Reviews. Molecular Cell Biology》 6 (11): 838–849. doi:10.1038/nrm1761. PMID 16261189. S2CID 31300025.
- ↑ Black JC, Van Rechem C, Whetstine JR (November 2012). “Histone lysine methylation dynamics: establishment, regulation, and biological impact”. 《Molecular Cell》 48 (4): 491–507. doi:10.1016/j.molcel.2012.11.006. PMC 3861058. PMID 23200123.
- ↑ Choudhary C, Kumar C, Gnad F, Nielsen ML, Rehman M, Walther TC, Olsen JV, Mann M (August 2009). “Lysine acetylation targets protein complexes and co-regulates major cellular functions”. 《Science》 325 (5942): 834–840. Bibcode:2009Sci...325..834C. doi:10.1126/science.1175371. PMID 19608861. S2CID 206520776.
- ↑ Shiio Y, Eisenman RN (November 2003). “Histone sumoylation is associated with transcriptional repression”. 《Proceedings of the National Academy of Sciences of the United States of America》 100 (23): 13225–13230. doi:10.1073/pnas.1735528100. PMC 263760. PMID 14578449.
- ↑ Wang H, Wang L, Erdjument-Bromage H, Vidal M, Tempst P, Jones RS, Zhang Y (October 2004). “Role of histone H2A ubiquitination in Polycomb silencing”. 《Nature》 431 (7010): 873–878. Bibcode:2004Natur.431..873W. doi:10.1038/nature02985. hdl:10261/73732. PMID 15386022. S2CID 4344378.
- ↑ 가 나 Shoulders MD, Raines RT (2009). “Collagen structure and stability”. 《Annual Review of Biochemistry》 78: 929–958. doi:10.1146/annurev.biochem.77.032207.120833. PMC 2846778. PMID 19344236.
- ↑ 가 나 Civitelli R, Villareal DT, Agnusdei D, Nardi P, Avioli LV, Gennari C (1992). “Dietary L-lysine and calcium metabolism in humans”. 《Nutrition》 8 (6): 400–405. PMID 1486246.
- ↑ 가 나 다 라 Vaz FM, Wanders RJ (February 2002). “Carnitine biosynthesis in mammals”. 《The Biochemical Journal》 361 (Pt 3): 417–429. doi:10.1042/bj3610417. PMC 1222323. PMID 11802770.
- ↑ Yamauchi M, Sricholpech M (2012년 5월 25일). “Lysine post-translational modifications of collagen”. 《Essays in Biochemistry》 52: 113–133. doi:10.1042/bse0520113. PMC 3499978. PMID 22708567.
- ↑ Vollmer W, Blanot D, de Pedro MA (March 2008). “Peptidoglycan structure and architecture”. 《FEMS Microbiology Reviews》 32 (2): 149–167. doi:10.1111/j.1574-6976.2007.00094.x. PMID 18194336.
- ↑ Curtiss R (May 1978). “Biological containment and cloning vector transmissibility”. 《The Journal of Infectious Diseases》 137 (5): 668–675. doi:10.1093/infdis/137.5.668. PMID 351084.
- ↑ 가 나 Flanagan JL, Simmons PA, Vehige J, Willcox MD, Garrett Q (April 2010). “Role of carnitine in disease”. 《Nutrition & Metabolism》 7: 30. doi:10.1186/1743-7075-7-30. PMC 2861661. PMID 20398344.
- ↑ 가 나 Chromiak JA, Antonio J (2002). “Use of amino acids as growth hormone-releasing agents by athletes”. 《Nutrition》 18 (7–8): 657–661. doi:10.1016/s0899-9007(02)00807-9. PMID 12093449.
- ↑ Corpas E, Blackman MR, Roberson R, Scholfield D, Harman SM (July 1993). “Oral arginine-lysine does not increase growth hormone or insulin-like growth factor-I in old men”. 《Journal of Gerontology》 48 (4): M128–M133. doi:10.1093/geronj/48.4.M128. PMID 8315224.
- ↑ Gaby AR (2006). “Natural remedies for Herpes simplex.”. 《Altern Med Rev》 11 (2): 93–101. PMID 16813459.
- ↑ Tomblin FA, Lucas KH (2001). “Lysine for management of herpes labialis.”. 《Am J Health Syst Pharm》 58 (4): 298–300, 304. doi:10.1093/ajhp/58.4.298. PMID 11225166.
- ↑ Chi CC, Wang SH, Delamere FM, Wojnarowska F, Peters MC, Kanjirath PP (2015년 8월 7일). “Interventions for prevention of herpes simplex labialis (cold sores on the lips)”. 《The Cochrane Database of Systematic Reviews》 (8): CD010095. doi:10.1002/14651858.CD010095.pub2. PMC 6461191. PMID 26252373.
- ↑ “Scientific Opinion on the substantiation of health claims related to L-lysine and immune defence against herpes virus (ID 453), maintenance of normal blood LDL-cholesterol concentrations (ID 454, 4669), increase in appetite leading to an increase in energ”. 《EFSA Journal》 9 (4): 2063. 2011. doi:10.2903/j.efsa.2011.2063. ISSN 1831-4732.
- ↑ Pinnell SR, Krane SM, Kenzora JE, Glimcher MJ (May 1972). “A heritable disorder of connective tissue. Hydroxylysine-deficient collagen disease”. 《The New England Journal of Medicine》 286 (19): 1013–1020. doi:10.1056/NEJM197205112861901. PMID 5016372.
- ↑ Rudman D, Sewell CW, Ansley JD (September 1977). “Deficiency of carnitine in cachectic cirrhotic patients”. 《The Journal of Clinical Investigation》 60 (3): 716–723. doi:10.1172/jci108824. PMC 372417. PMID 893675.
- ↑ 가 나 Rushton DH (July 2002). “Nutritional factors and hair loss”. 《Clinical and Experimental Dermatology》 27 (5): 396–404. doi:10.1046/j.1365-2230.2002.01076.x. PMID 12190640. S2CID 39327815.
- ↑ Emery PW (October 2005). “Metabolic changes in malnutrition”. 《Eye》 19 (10): 1029–1034. doi:10.1038/sj.eye.6701959. PMID 16304580.
- ↑ Ghosh S, Smriga M, Vuvor F, Suri D, Mohammed H, Armah SM, Scrimshaw NS (October 2010). “Effect of lysine supplementation on health and morbidity in subjects belonging to poor peri-urban households in Accra, Ghana”. 《The American Journal of Clinical Nutrition》 92 (4): 928–939. doi:10.3945/ajcn.2009.28834. PMID 20720257.
- ↑ 가 나 Houten SM, Te Brinke H, Denis S, Ruiter JP, Knegt AC, de Klerk JB, Augoustides-Savvopoulou P, Häberle J, Baumgartner MR, Coşkun T, Zschocke J, Sass JO, Poll-The BT, Wanders RJ, Duran M (April 2013). “Genetic basis of hyperlysinemia”. 《Orphanet Journal of Rare Diseases》 8: 57. doi:10.1186/1750-1172-8-57. PMC 3626681. PMID 23570448.
- ↑ Hoffmann, Georg F.; Kölker, Stefan (2016). 《Inborn Metabolic Diseases》. Springer, Berlin, Heidelberg. 333–348쪽. doi:10.1007/978-3-662-49771-5_22. ISBN 978-3-662-49769-2.
- ↑ Dancis J, Hutzler J, Ampola MG, Shih VE, van Gelderen HH, Kirby LT, Woody NC (May 1983). “The prognosis of hyperlysinemia: an interim report”. 《American Journal of Human Genetics》 35 (3): 438–442. PMC 1685659. PMID 6407303.
- ↑ Mills PB, Struys E, Jakobs C, Plecko B, Baxter P, Baumgartner M, Willemsen MA, Omran H, Tacke U, Uhlenberg B, Weschke B, Clayton PT (March 2006). “Mutations in antiquitin in individuals with pyridoxine-dependent seizures”. 《Nature Medicine》 12 (3): 307–309. doi:10.1038/nm1366. PMID 16491085. S2CID 27940375.
- ↑ Mills PB, Footitt EJ, Mills KA, Tuschl K, Aylett S, Varadkar S, Hemingway C, Marlow N, Rennie J, Baxter P, Dulac O, Nabbout R, Craigen WJ, Schmitt B, Feillet F, Christensen E, De Lonlay P, Pike MG, Hughes MI, Struys EA, Jakobs C, Zuberi SM, Clayton PT (July 2010). “Genotypic and phenotypic spectrum of pyridoxine-dependent epilepsy (ALDH7A1 deficiency)”. 《Brain》 133 (Pt 7): 2148–2159. doi:10.1093/brain/awq143. PMC 2892945. PMID 20554659.
- ↑ Hagen J, te Brinke H, Wanders RJ, Knegt AC, Oussoren E, Hoogeboom AJ, Ruijter GJ, Becker D, Schwab KO, Franke I, Duran M, Waterham HR, Sass JO, Houten SM (September 2015). “Genetic basis of alpha-aminoadipic and alpha-ketoadipic aciduria”. 《Journal of Inherited Metabolic Disease》 38 (5): 873–879. doi:10.1007/s10545-015-9841-9. PMID 25860818. S2CID 20379124.
- ↑ Hedlund GL, Longo N, Pasquali M (May 2006). “Glutaric acidemia type 1”. 《American Journal of Medical Genetics Part C: Seminars in Medical Genetics》 142C (2): 86–94. doi:10.1002/ajmg.c.30088. PMC 2556991. PMID 16602100.
- ↑ “Hyperlysinuria | Define Hyperlysinuria at Dictionary.com”.
- ↑ 가 나 Walter, John; John Fernandes; Jean-Marie Saudubray; Georges van den Berghe (2006). 《Inborn Metabolic Diseases: Diagnosis and Treatment》. Berlin: Springer. 296쪽. ISBN 978-3-540-28783-4.
- ↑ 가 나 “Norwegian granted for improving lysine production process”. 《All About Feed》. 2010년 1월 26일. 2012년 3월 11일에 원본 문서에서 보존된 문서.
- ↑ Toride Y (2004). 〈Lysine and other amino acids for feed: production and contribution to protein utilization in animal feeding〉. 《Protein sources for the animal feed industry; FAO Expert Consultation and Workshop on Protein Sources for the Animal Feed Industry; Bangkok, 29 April - 3 May 2002》. Rome: Food and Agriculture Organization of the United Nations. ISBN 978-92-5-105012-5.
- ↑ Abelson PH (March 1999). “A potential phosphate crisis”. 《Science》 283 (5410): 2015. Bibcode:1999Sci...283.2015A. doi:10.1126/science.283.5410.2015. PMID 10206902. S2CID 28106949.
- ↑ Coyne, Jerry A. (1999년 10월 10일). “The Truth Is Way Out There”. 《The New York Times》. 2008년 4월 6일에 확인함.
- ↑ Wu G (May 2009). “Amino acids: metabolism, functions, and nutrition”. 《Amino Acids》 37 (1): 1–17. doi:10.1007/s00726-009-0269-0. PMID 19301095. S2CID 1870305.
- ↑ Connor JM (2008). 《Global Price Fixing》 2판. Heidelberg: Springer-Verlag. ISBN 978-3-540-78669-6.
- ↑ Eichenwald, Kurt (2000). 《The Informant: a true story》. New York: Broadway Books. ISBN 978-0-7679-0326-4.