Influenzaviren
| Influenzaviren | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
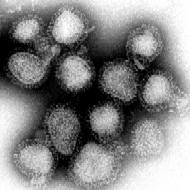
Influenzavirus A/Hong Kong/1/68 bei 70.000-facher Vergrößerung | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Alphainfluenzavirus bis Deltainfluenzavirus | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Influenzaviren sind Viren aus der Familie der Orthomyxoviridae, welche die Krankheit Influenza auslösen können. Sie sind behüllte[3] Viren mit einzelsträngigen, segmentierten RNA von negativer Polarität als Genom.
Zu den Influenzaviren zählen die Gattungen Alpha-, Beta-, Gamma- und Deltainfluenzavirus. Unter den Gattungen finden sich auch die Erreger der Influenza oder „echten“ Grippe. Gemäß International Committee on Taxonomy of Viruses (ICTV, Stand: 30. März 2024) haben alle vier Gattungen jeweils nur eine Spezies, die mit dem Art-Epitheton influenzae bezeichnet wird. Diesen vier umfassen jeweils nur ein Virus, und zwar der Reihe nach das Influenza-A-Virus (FLUAV oder IAV) bis Influenza-D-Virus (FLUDV oder IDV).[4]
Aufbau
[Bearbeiten | Quelltext bearbeiten]
Virion
[Bearbeiten | Quelltext bearbeiten]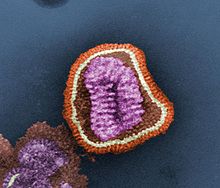
Das innerhalb der Lipidhülle (genauer: Virusmembran) befindliche Ribonukleoprotein des Virusteilchens (Virions), besitzt eine ungefähr helikale Symmetrie. Das Ribonukleoprotein ist ein Komplex aus dem Genom des Virus, den strukturellen (M1 und NP) und den replikationsrelevanten Proteinen (PA, PB1, PB2).
Im Transmissionselektronenmikroskop sieht man alle Gattungen dieses Virus als kugelige oder sphärisch ellipsoide (rundliche bis eiförmige), gelegentlich auch filamentöse (fadenförmige), umhüllte Viruspartikel mit einem Durchmesser von 80 bis 120 nm,[5] in deren Lipidhülle eine variierende Anzahl der drei Membranproteine HA, NA und M2 beim Influenza-A-Virus bzw. der zwei Membranproteine HA und NA beim Influenza-B-Virus (IBV) oder der Hämagglutinin-Esterase-Faktor HEF und das Matrixprotein CM2 beim Influenza-C-Virus (ICV) eingelagert sind. Die Glykoproteine HA und NA ragen als 10 bis 14 nm lange Spikes oder Peplomere genannte Fortsätze über die Virusoberfläche des Influenza-A-Virus (IAV) hinaus. Dagegen ragt das M2 des Influenza-A-Virus nur mit 24 Aminosäuren aus der Lipiddoppelschicht heraus und wird unter der Überdeckung des HA und NA kaum von Antikörpern im Zuge einer Immunreaktion erkannt. Beim Influenza-A- und Influenza-B-Virus sind daher genau zwei Typen dieser Spikes serologisch von besonderem Interesse: das Hämagglutinin (HA) und die Neuraminidase (NA), gegen die Antikörper nach einer Erkrankung und in geringerem Umfang auch nach einer Impfung mit einem Influenzaimpfstoff gegen Influenza A und B entstehen. Diese Antikörper können zur serologischen Klassifizierung der 18 HA- und 11 NA-Subtypen von Influenza-A-Viren herangezogen werden (nach aktuellem Stand 2017, siehe hierzu auch A/H18N11).
Genom
[Bearbeiten | Quelltext bearbeiten]Das Genom der Influenzaviren besteht gewöhnlich aus acht RNA-Abschnitten (Segmenten) negativer Polarität, bei den selteneren Influenza-C- und -D-Viren sind es nur sieben. Diese RNA-Moleküle enthalten die genetische Information, die für die Vermehrung und den Zusammenbau der Viruspartikel benötigt wird und kommen im Virion bevorzugt einzelsträngig vor.[5] Ebenso kommt in einem Virion nur eine Kopie des Genoms vor.[6] Die Segmentierung des Genoms ist auch für die erhebliche Steigerung der genetischen Veränderlichkeit (Variabilität) der Influenzaviren über die Fähigkeit zur genetischen Reassortierung (auch Antigen-Shift) verantwortlich, da bei einer Superinfektion einer Zelle (mit einem anderen Influenzastamm) ein Austausch der Segmente erfolgen kann. Durch das RNA-basierte einzelsträngige Genom kommen häufig Punktmutationen vor (sogenannter Antigen-Drift), denn die RNA-Polymerasen von RNA-Viren besitzen keine Exonuklease-Funktion zur Korrektur von Kopierfehlern. Durch beide Mechanismen entstehen Fluchtmutationen zur Umgehung der Immunantwort, während die Funktionen des Virions erhalten bleiben sollen. Das Genom erhält die stärkste Anpassung durch den vom Immunsystem ihrer Reservoirwirte erzeugten Selektionsdruck, wobei der Erhalt der Funktionen der Proteine für eine hohe Reproduktions- und Infektionsrate notwendig ist.
Die acht verschiedenen RNA-Segmente (HA, NA, M, NP, PA, PB1, PB2 und NS) kodieren beim Influenza-A-Virus üblicherweise zehn, gelegentlich elf virale Proteine:[7] das Hämagglutinin (HA), die Neuraminidase (NA), das Nukleoprotein (NP), die Matrixproteine (M1) und (M2), die RNA-Polymerase (PA), die Polymerase-bindenden Proteine (PB1, PB2 und vereinzelt auch PB1-F2) und die Nichtstrukturproteine (NS1 und NS2). Beim Genom des Influenza-C-Virus mit seinen sieben Segmenten fehlt die Neuraminidase, da deren Funktion mit der Hämagglutinin-Funktion im HEF integriert ist. Aus den RNA-Segmenten M, NS und in manchen Stämmen auch aus PB1 entstehen beim Influenza-A-Virus durch alternatives Spleißen jeweils zwei Proteine, M1 und M2 bzw. NS1 und NS2 bzw. PB1 und PB1-F2. Am 5'- und am 3'-Ende jedes Segments befindet sich im Virion ein Polymerasekomplex aus PA, PB1 und PB2.[8]
Hüllproteine
[Bearbeiten | Quelltext bearbeiten]

Das Hämagglutinin (HA) ist ein Lektin und bewirkt die Verklumpung (Agglutination) von Erythrozyten und vermittelt bei der Infektion die Anheftung des Virusteilchens (Virions) an eine Wirtszelle. Das Ankoppeln des Hämagglutinins an eine Zelle geschieht durch eine Anlagerung eines Teils des Hämagglutininmoleküls an Sialinsäuren (SA) auf Proteinen der Wirtszellenhülle, die als Rezeptoren (SA-Rezeptoren) fungieren.[9] Diese Sialinsäuren sind bei Vögeln gehäuft α2,3-verknüpft und bei Säugern gehäuft α2,6-verknüpft.[10] Daneben unterscheiden sich die Verteilungen dieser Verknüpfungen in der Lunge in Säugern und Vögeln.[11] Jede Hämagglutininvariante passt dabei an einen bestimmten Wirtszellenrezeptor nach dem Schlüssel-Schloss-Prinzip, wobei jeder Wirt nur über einen Teil aller von Influenzaviren genutzten Rezeptoren verfügt. Diese Tatsache ist auch der Grund dafür, dass bestimmte Subtypen oder Virusvarianten mit ihrem speziellen Hämagglutinintyp bestimmte Wirte leicht infizieren und dabei eine Erkrankung auslösen können und andere prinzipiell mögliche Wirte wiederum nicht oder nur sehr eingeschränkt. Nach einer proteolytischen Aktivierung des von einer Zelle aufgenommenen Hämagglutinins durch zelluläre Serinpeptidasen und einer Ansäuerung des Endosoms bewirkt das Hämagglutinin in seiner zweiten Funktion als fusogenes Protein über seine Fusionsdomäne eine Fusion mit der endosomalen Membran zur Freisetzung des Ribonukleoproteins ins Zytosol. Das Hämagglutinin ist aus drei gleichen Proteinen aufgebaut (ein Homotrimer). Mittels Proteolyse wird jeder dieser drei Teile (Monomere) wiederum in zwei Polypeptidketten gespalten, die jedoch über eine Disulfidbrücke miteinander verbunden bleiben. Diese Spaltung ist bei der Entpackung der Viren für die Fusion der Virusmembran mit der Endosomenmembran zwingend notwendig, nicht jedoch für die Rezeptorbindung. Durch Mutationen, besonders in Hinblick auf mögliche Veränderungen des Hämagglutinins, kann sich die Infektionsgefahr für den einen oder anderen potentiellen Wirt erheblich ändern. Allerdings können die Viren die HA-Bindungsstelle nicht beliebig verändern, da bei einem Funktionsverlust der Wiedereintritt in Zellen nicht mehr erfolgen kann und somit die Infektkette unterbrochen wird.[10] Vor allem gegen das Hämagglutinin werden neutralisierende Antikörper ausgebildet, die eine erneute Infektion mit demselben Virusstamm verhindern. Daher sind neu auftretende Epidemien meistens von Änderungen im Hämagglutinin begleitet.[12]
Die Neuraminidase (NA) hat im Infektionsvorgang viele Funktionen, darunter eine enzymatische Funktion zur Abspaltung (Hydrolyse) der N-Acetylneuraminsäure (eine Sialinsäure) an zellulären Rezeptoren. Dadurch erfolgt die Freisetzung der durch die Replikation neu entstandenen Viren (Tochtervirionen aus den infizierten Zellen) und damit eine Ausbreitung der Infektion sowohl innerhalb desselben Organismus als auch auf andere Organismen.[13] Außerdem verhindert die Neuraminidase ein Hämagglutinin-vermitteltes Anheften der Tochtervirionen an bereits infizierte Zellen, weil die infizierten Zellen durch die Neuraminidase auf ihrer Zelloberfläche kaum noch N-Acetylneuraminsäure auf ihrer Zelloberfläche tragen. Als Nebeneffekt wird der Schleim in der Lunge verflüssigt. Daneben verhindert die Neuraminidase, dass in einer infizierten Zelle während der Replikation ein Zelltodprogramm gestartet werden kann.[14][15] Oseltamivir, Zanamivir und Peramivir hemmen bei nicht-resistenten Influenza-A- und Influenza-B-Varianten die Neuraminidase.
Das Matrixprotein 2 (M2) ist das kleinste der drei Membranproteine des Influenza-A-Virus. Bei diesem besteht M2 aus circa 97 Aminosäuren, wovon 24 Aminosäuren aus der Membran herausragen. Das Matrixprotein M2 ist bei Influenza-A-Viren ein Protonenkanal zur Ansäuerung des Inneren des Virions nach einer Endozytose, so dass die Fusionsdomäne des Hämagglutinins ausgelöst wird und eine Verschmelzung von Virus- und Endosomenmembran zur Freisetzung des Ribonukleoproteins ins Zytosol erfolgen kann.[6] Gleichzeitig bewirkt die Ansäuerung im Inneren des Virions eine Dissoziation des Matrixproteins 1 vom Ribonukleoprotein.[6] Beim Influenza-B-Virus ist das Matrixprotein 2 (BM2) kein Hüllprotein, sondern ein lösliches Protein von circa 109 Aminosäuren.[16] Beim Influenza-C-Virus ist das entsprechende Matrixprotein CM2, wie beim Influenza-A-Virus, ein Ionenkanal. Amantadin und Rimantadin hemmen bei nicht-resistenten Influenza-A-Varianten das Matrixprotein M2.
Interne Proteine
[Bearbeiten | Quelltext bearbeiten]Als weitere im Virion befindliche Proteine (Strukturproteine, englisch structural proteins) existieren neben den Hüllproteinen die internen Proteine (engl. core proteins). Der Bereich zwischen Virusmembran und Ribonukleoprotein wird als Matrix oder Viruslumen (lateinisch lumen für ‚Licht‘) bezeichnet, da er im Transmissionselektronenmikroskop aufgrund einer niedrigeren Elektronendichte heller als das Ribonukleoprotein erscheint. An der Innenseite der Virusmembran befinden sich die zytosolischen Anteile der drei Membranproteine HA, NA und M2, sowie das Matrixprotein M1, welches die Virusmembran mit dem Ribonukleoprotein verbindet, bei Ansäuerung aber das Ribonukleoprotein freisetzt. Das Ribonukleoprotein besteht aus dem Nucleoprotein NP, den viralen RNA-Segmenten und den für die Replikation und Transkription notwendigen Proteinen des Polymerase-Komplexes (PA, PB1 und PB2, mit A für acide oder B für basisch bezeichnet, je nach ihren isoelektrischen Punkten).[17] Das Nucleoprotein NP bindet neben dem Matrixprotein M1 an die virale RNA und vermittelt über sein Kernlokalisierungssignal den Transport in den Zellkern.[18] NS2 kommt in geringen Mengen gelegentlich auch im Virion vor.[19]
Nichtstrukturproteine
[Bearbeiten | Quelltext bearbeiten]Die regulatorischen Proteine NS1, NS2 und das bei manchen Stämmen auftretende PB1-F2 kommen nicht im Virion vor und werden daher als Nichtstrukturproteine bezeichnet. Das NS1 mindert durch seine Bindung an PDZ-Domänen die Interferon-Reaktion des Wirtes und somit die Immunreaktion.[20] Daneben verhindert NS1 das Ausschleusen wirtseigener mRNA aus dem Zellkern durch Bindung ihrer Cap-Struktur, wodurch die virale RNA vermehrt in Proteine translatiert werden.[21] NS2 vermittelt den Export viraler mRNA aus dem Zellkern.[22] PB1-F2 fördert die korrekte Lokalisation des PB1 im Zellkern und bindet an die Polymerase PA und fördert über den mitochondrialen Weg die Einleitung der Apoptose zur Verbesserung der Freisetzung der Tochtervirionen.[23]
Wirtsrestriktion
[Bearbeiten | Quelltext bearbeiten]Die Koevolution von Menschen und Viren (in diesem Falle von RNA-Viren) hat im Menschen antivirale Mechanismen hervorgebracht, die als Wirtsrestriktions- oder auch Resistenzfaktoren (engl. host restriction factors) bezeichnet werden. Hierzu zählen bei Influenzaviren der Myxovirus-Resistenzfaktor Mx1,[24] NOD-2,[25] die Toll-like Rezeptoren 3,[26] 7[27] und 8,[28] RIG-I,[26] der dsRNA-aktivierte Inhibitor der Translation DAI,[29] MDA5,[30] die Oligoadenylatsynthase OAS1,[31] das Nod-like Receptor Protein 3 (NLRP-3)[32] und die Proteinkinase R.[33]
Replikationszyklus
[Bearbeiten | Quelltext bearbeiten]
Für den Replikationszyklus des Influenza-A-Virus sind mindestens 219 wirtseigene Proteine notwendig.[34]
Import
[Bearbeiten | Quelltext bearbeiten]Die Influenzaviren werden beim Menschen im Atemtrakt (Respirationstrakt) eines infizierten Individuums repliziert. Menschliche Grippeviren bevorzugen Zellen des Flimmerepithels. Im Gegensatz dazu vermehrt sich das Grippe-Virus bei Vögeln hauptsächlich in den Darmepithelzellen.[35]
Die Viren wandern bei ihrem Wirt durch das Mucin (Schleim) in die Epithelzellen, die als Wirtszellen dienen. Dafür, dass sie dabei nicht mit dem Schleim verkleben, sorgt die Neuraminidase, welche den Schleim verflüssigt.[36] Nach der Bindung des Hämagglutinins an eine N-Acetyl-Neuraminsäure auf einer Zelloberfläche erfolgt die Einstülpung des Virions durch Endozytose. Im Endosom schneiden zelluläre Serinproteasen das Hämagglutinin in seine aktivierte Form. Daneben sinkt der pH-Wert im Endosom, wodurch über das Ionenkanalprotein M2 das Innere des Virions angesäuert wird. Die Ansäuerung löst die Fusionsdomäne des Hämagglutinins aus, wodurch die Virusmembran mit der Endosomenmembran verschmilzt und das Ribonukleoprotein ins Zytosol freigesetzt wird, gleichzeitig löst sich M1 vom Ribonukleoprotein, wodurch die Kernlokalisierungssignale des NP im Ribonukleoprotein exponiert werden. Das Ribonukleoprotein wird auch durch einen zellulären Abbaumechanismus, das Aggregosom, vom Matrixprotein befreit, vermutlich unter Verwendung von zelleigenen Motorproteinen.[37] Über die Kernlokalisierungssequenz des Nukleoproteins erfolgt anschließend ein Import des Ribonukleoproteins in den Zellkern.[6]
Replikation
[Bearbeiten | Quelltext bearbeiten]Die Kopie der viralen RNA erfolgt bei IAV und IBV durch den viralen Polymerasekomplex – ein Heterotrimer aus den drei Proteinen PA, PB1 und PB2 – unter Verwendung der Ribonukleotide des Wirts.[38] Die Transkription zur Erzeugung der viralen mRNA erfolgt durch selten vorkommenden Mechanismus des Cap snatching.[39] Die mit 5'-methylierten Cap-Strukturen modifizierten zellulären mRNA werden von PB2 an der 7-Methylguanosingruppe gebunden und anschließend 10 bis 13 Nukleotide nach der Cap-Struktur durch PA geschnitten. Die kurzen Cap-tragenden Fragmente werden von PB1 gebunden und im RNA-Polymerase-Komplex als Primer für die Transkription viraler RNA verwendet. Der Polymerase-Komplex bindet dabei kurzfristig an die zelluläre RNA-Polymerase II.[39] Nebenbei wird dadurch die mRNA des Wirts zerlegt, so dass das Ribosom freier für die Synthese der viralen mRNA ist. Während die zelluläre mRNA eine Cap-Struktur und eine Polyadenylierung trägt, hat die virale RNA der Replikation beides nicht.[40]
Export
[Bearbeiten | Quelltext bearbeiten]Durch das NS2-Protein wird die vervielfältigte virale RNA aus dem Zellkern ins Zytosol geschleust, wo nach der RNA-Vorlage am Ribosom Proteine hergestellt werden. Die strukturellen Proteine binden aneinander, der Polymerase-Komplex aus PA, PB1 und PB2 bindet an das 5'- und das 3'-Ende der viralen RNA und das NP-Protein bindet an die restliche virale RNA, wodurch sich das Ribonukleoprotein zusammenfügt.[39][41] Die viralen Hüllproteine HA und NA sammeln sich an der Zelloberfläche an den Lipid Rafts, nicht aber M2.[6] Das Ribonukleoprotein bindet an die Innenseite der Lipid Rafts. Der Mechanismus der Knospung ist noch nicht geklärt.[6] Influenzaviren sind lytische Viren. Sie induzieren einen programmierten Zelltod der Wirtszelle.[42]
In einer einzigen infizierten Wirtszelle können sich bis zu etwa 20.000 neue Influenzaviren bilden (englisch burst size ‚Berstgröße‘), bevor diese dann abstirbt und anschließend die freigesetzten Viren weitere Nachbarzellen infizieren. In infizierten Zellkulturen oder embryonierten Hühnereiern wurden virusstammabhängige Werte zwischen 1.000 und 18.755 Tochtervirionen pro Zelle ermittelt.[43][44][45] Diese produzieren dann ebenfalls jeweils viele Tausende neuer Viren. So erklärt sich auch die Schnelligkeit, mit der sich in der Regel diese virale Infektion im Körper eines Betroffenen ausbreitet.
Systematik
[Bearbeiten | Quelltext bearbeiten]Es gibt vier verschiedene Gattungen der Influenzaviren (Alpha bis Delta), welche mit weiteren Gattungen wie Isavirus, Quaranjavirus, Thogotovirus etc. zusammen zur Familie der Orthomyxoviren (Orthomyxoviridae) gehören.
In Fachkreisen wird jede Virusvariante mit den Kennungen Typus, Ort der erstmaligen Isolierung (Virusanzucht), Nummer des Isolats, Isolierungsjahr (Beispiel: Influenza B/Shanghai/361/2002) und nur bei den A-Viren auch zusätzlich mit der Kennung des Oberflächenantigens benannt [Beispiel: Influenza A/California/7/2004 (H3N2)].[46] Ebenso werden Influenzaviren nach ihrem natürlichen Wirt benannt (Vogelgrippeviren, Schweinegrippeviren).[47] Allerdings werden die ausgelösten Grippen nur als Vogelgrippen oder Schweinegrippe bezeichnet, wenn die Infektion in dieser Wirtsspezies stattfindet, nicht aber beim Menschen.[47] Wenn der Mensch aufgrund einer Veränderung eines Influenzavirus zur häufiger infizierten Wirtsspezies wird, verwendet man die Schreibweise der Serotypen mit einem nachgestellten v (von engl. variant), z. B. H1N1v, H3N2v.[48]
Alphainfluenzavirus mit Spezies A. influenzae
[Bearbeiten | Quelltext bearbeiten]
- Die lineare, einzelsträngige RNA ihres Genoms besitzt acht Segmente und sie zeichnen sich besonders durch große Unterschiede in den antigenen Eigenschaften aus, die im Vergleich zu den anderen Gattungen auf besonders hoher Mutationsfrequenz und Neugruppierungen beruhen. Diese Untertypen befallen üblicherweise jeweils nur bestimmte Wirte und innerhalb derer bestimmte Zellarten (siehe Tropismus). Dazu zählen der Mensch und verschiedene Säugetierarten wie Schweine (siehe unter Schweineinfluenza), Pferde (siehe unter Pferdeinfluenza), Nerze, Seehunde und Wale, Haushunde, einige Katzenarten und zahlreiche Vogelarten.[49] Das primäre Reservoir aller Influenza-A-Subtypen liegt im Wassergeflügel.[50] Alle der ersten 16 HA- und 9 NA-Serotypen von Influenza A können Vögel infizieren.[51] Die erst 2012 und 2013 entdeckten Subtypen A/H17N10 und A/H18N11 wurden hingegen bisher nur aus Fruchtfledermäusen isoliert.
- Neuere Forschungen konnten auch Wirkungen der Influenza-A-Viren auf die Hämatopoese belegen.[52]
Influenza-A-Subtypen
[Bearbeiten | Quelltext bearbeiten]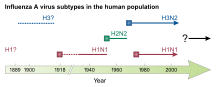


Im Allgemeinen werden die Subtypen der Spezies Alphainfluenzavirus influenzae (mit Influenza-A-Virus, FLUA oder IAV) in erster Linie serologisch eingeteilt. Dies geschieht nach dem Muster A/HxNy oder A/Isolierungsort/Isolat/Jahr (HxNy). Eine große Anzahl von Untertypen sind identifiziert.[53]
Die wichtigsten Oberflächenantigene beim Influenza-A-Virus für die Infektion des Menschen sind die Hämagglutinin-Serotypen H1, H2, H3, H5, seltener H7 und H9 und die Neuraminidase-Serotypen N1, N2, seltener N7, weshalb auch folgende Subtypen für den Menschen von besonderer Bedeutung sind:
- Influenza-A-Virus H1N1
- Eine Variante von Influenza A/H1N1 konnte als Auslöser der so genannten Spanischen Grippe von 1918/1920 im Lungengewebe von Opfern nachgewiesen werden. 2005 gelang Jeffery Taubenberger eine Rekonstruktion des Erregers der Spanischen Grippe aus Genfragmenten. Ein erneuter weltweiter Ausbruch – die so genannte Russische Grippe – ereignete sich 1977.[54] Im April 2009 ereignete sich in Mexiko ein epidemieartiger Ausbruch einer bis dahin unbekannten Variante des H1N1-Subtyps, an dem zahlreiche Menschen erkrankten (siehe: Pandemie H1N1 2009/10). Da der erste Nachweis von A/H1N1 im Jahr 1930 aus Schweinen erfolgte,[55][56] werden durch diesen Subtyp verursachte Infektionen beim Schwein als Schweineinfluenza bezeichnet.
- Influenza-A-Virus H2N2
- Ein weltweiter Ausbruch dieses Subtyps der Humaninfluenza war 1957 die Ursache einer Pandemie, die als Asiatische Grippe bezeichnet wurde.
- Influenza-A-Virus H3N2
- Ein weltweiter Ausbruch dieses Subtyps (A/Hong Kong/1/1968 H3N2)[57] war 1968 die Ursache einer Pandemie, die als Hongkong-Grippe bezeichnet wurde.
- Influenza-A-Virus H5N1
- Dieser Subtyp ist bislang nur in sehr seltenen Einzelfällen von Mensch zu Mensch übertragbar, gleichwohl gab es seit 2003 mehrere hundert von der WHO gemeldete Todesfälle (→ Vogelgrippe H5N1).
- Influenza-A-Virus H7N9
- Vermutlich nach Kontakt mit infiziertem Geflügel kam es im Februar 2013 erstmals beim Menschen zu Infektionen mit dem Influenza-A-Virus H7N9 und als deren Folgen zu Todesfällen durch die sogenannte Vogelgrippe H7N9 aufgrund schwerer Pneumonien durch eine bis dahin unbekannte, reassortierte Variante des Virus A/H7N9.[58] In seltenen Fällen ist eine Übertragung von Mensch zu Mensch möglich. Allerdings ist der Erreger nur begrenzt übertragbar und es fand bislang keine anhaltende Mensch-zu-Mensch-Übertragung statt.[59][60]
Informationen zu Influenza-A-Subtypen bei Vögeln finden sich im Artikel Geflügelpest.
Betainfluenzavirus mit Spezies B. influenzae
[Bearbeiten | Quelltext bearbeiten]- Ihr Genom hat ebenfalls eine acht-fach segmentierte lineare, einzelsträngige RNA und sie befallen nur Menschen[61] und Seehunde.[62][63]
Influenza-B-Subtypen
[Bearbeiten | Quelltext bearbeiten]Die Spezies Betainfluenzavirus influenzae (mit Influenza-B-Virus, FLUB oder IBV) wird nach dem Ort des Auftretens in mehrere Stamm-Linien eingeteilt, z. B.:[64]
- B/Victoria-Linie[65]
- B/Yamagata-Linie[66]
- B/Yamaguchi-Linie[67]
- B/Yokohama-Linie[68]
- B/Yunnan-Linie[69]
- B/Zhuhai-Linie[70]
Gammainfluenzavirus mit Spezies G. influenzae
[Bearbeiten | Quelltext bearbeiten]- Im Gegensatz zu den Influenza-A- und B-Viren hat die lineare, einzelsträngige RNA des Genoms der Influenza-C-Viren nur sieben Segmente und sie besitzen keine Neuraminidase (NA). Außerdem liegt bei diesen Viren ein Oberflächen-Glykoprotein Haemagglutinin-Esterase-Fusion-Protein (HEF) vor, das sowohl die Aufgaben der Rezeptorbindung des Virus an die Wirtszelle, der anschließenden Eindringung (Fusion) wie auch der späteren Freisetzung der neugebildeten Viren aus der Zelle übernimmt. Das Influenza-C-Virus befällt Menschen und Schweine (siehe ebenfalls unter Schweineinfluenza), doch spielt er bei Erkrankungen des Menschen keine relevante Rolle, da es, wenn überhaupt, nur zu milden Erkrankungen führt.
Influenza-C-Subtypen
[Bearbeiten | Quelltext bearbeiten]Die Unterschiede zwischen einzelnen Virusstämmen der Spezies Gammainfluenzavirus influenzae (mit Influenza-C-Virus, FLUC oder ICV) sind gering. Eine Unterteilung in Subtypen findet sich beim NCBI.[71]
Deltainfluenzavirus mit Spezies D. influenzae
[Bearbeiten | Quelltext bearbeiten]- Vom ICTV mit Stand 2018 bestätigte weitere Variante.
Influenza-D-Subtypen
[Bearbeiten | Quelltext bearbeiten]Die ersten Vertreter der Spezies Deltainfluenzavirus influenzae (mit Influenza-D-Virus, FLUD oder IDV) wurden 2011 isoliert.[72] Diese Gattung scheint sehr nahe mit Influenza C verwandt zu sein, die Aufspaltung erfolgte offenbar erst vor einigen hundert Jahren,[73] das Genom hat daher ebenfalls sieben Segmente.[74] Es gibt gegenwärtig mindestens zwei Subtypen.[75] Hauptsächlich werden Rinder infiziert, aber auch Schweine. Eine Unterteilung in Subtypen findet sich beim NCBI.[76]
Variabilität
[Bearbeiten | Quelltext bearbeiten]Antigendrift
[Bearbeiten | Quelltext bearbeiten]
Eine Häufung von Punktmutationen in den Nukleotiden führt zu einer Veränderung der Erbinformationen (Gendrift). Die Punktmutationen entstehen bei Influenzaviren hauptsächlich durch die Ungenauigkeit des Polymerase-Komplexes. Betreffen solche Veränderungen die beiden Glykoproteine HA und NA (bzw. den sie kodierenden Bereich), bewirkt dies eine Änderung der Oberflächenantigene des Grippevirus (Antigendrift). Menschliche Antikörper und cytotoxische T-Zellen können immer nur eine solche Variante erkennen, ein nun unerkanntes Virus wird als Fluchtmutante bezeichnet. Diese eher kleinen Veränderungen sind der Grund dafür, dass ein Mensch mehrmals in seinem Leben mit einer anderen, nur geringfügig veränderten Virusvariante (Driftvariante) infiziert werden kann und dass sowohl Epidemien wie regional begrenzte Ausbrüche regelmäßig wiederkehren.
Daher ist es ein Ziel des Influenza-Impfstoffdesigns, breitneutralisierende Antikörper hervorzurufen. Nach mehreren überstandenen Infektionen mit unterschiedlichen Stämmen kommt es langfristig zu einem Anstieg der Titer von breitneutralisierenden Anti-Influenza-Antikörpern, möglicherweise aufgrund der hohen genetischen Variabilität der Influenzavirusstämme.[77] Ebenso wird versucht, die zelluläre Immunantwort auf konservierte Bereiche der viralen Proteine zu lenken, bei denen weniger Fluchtmutationen entstehen können. Die Mutationsrate des Influenzavirus A liegt bei 0,000015 Mutationen pro Nukleotid und Replikationszyklus.[78] Allerdings beeinflusst das bei einer Immunreaktion gebildete Repertoire an Antikörpern und zytotoxischen T-Zellen prägend ihre jeweilige Immundominanz, was die Anpassung der Immunreaktion bei späteren Infektionen mit veränderten Influenzaviren beeinflusst (Antigenerbsünde).
Antigenshift
[Bearbeiten | Quelltext bearbeiten]
Wird ein Organismus gleichzeitig von zwei Virusvarianten infiziert (Doppelinfektion) oder es tauschen zwei Viren gleichen Ursprungs unterschiedlich mutierte Gensegmente aus (wobei im letzteren Fall die Veränderungen geringer ausfallen), so kann es zu einer Neuzusammenstellung unter den je acht Genomsegmenten der beteiligen Influenzaviren kommen, in dem einzelne oder mehrere RNA-Moleküle zwischen den Influenzaviren in einer doppelt infizierten Zelle ausgetauscht werden. Diesen Vorgang nennt man genetische Reassortierung, und sie kann im Menschen, aber auch in anderen Wirten, wie beispielsweise bei Vögeln und Schweinen erfolgen. Die so verursachten größeren, als Antigenshift bezeichnete Veränderungen in den viralen Oberflächenantigenen werden allein beim Influenza-A-Virus beobachtet (Shiftvarianten), allerdings kommen sie nur selten vor. Derartige Veränderungen können dann der Ursprung von Pandemien sein, von denen es im 20. Jahrhundert die von 1918 bis 1919 mit dem Influenza-A-Subtyp H1N1, 1957 mit H2N2, 1968 mit H3N2 und die von 1977 mit dem Wiederauftauchen von H1N1 gab.
Umweltstabilität
[Bearbeiten | Quelltext bearbeiten]Je nach Temperatur ist die Umweltstabilität (synonym Tenazität) der Influenzaviren sehr unterschiedlich. Bei einer normalen sommerlichen Tagestemperatur von etwa 20 °C können an Oberflächen angetrocknete Viren in der Regel zwei bis acht Stunden überdauern. Bei einer Temperatur von 0 °C mehr als 30 Tage und im Eis sind sie nahezu unbegrenzt überdauerungsfähig. Bei 22 °C überstehen sie sowohl in Exkrementen wie auch in Geweben verstorbener Tiere und in Wasser mindestens vier Tage. Die Lipidhülle des Influenza-Virus verändert sich bei tieferen Temperaturen, wodurch das Virus stabilisiert wird und länger pathogen bleibt.[79] Oberhalb von 22 °C verringert sich allerdings die Umweltstabilität der Influenzaviren sehr deutlich. Bei 56 °C werden sie innerhalb von 3 Stunden und bei 60 °C innerhalb von 30 Minuten inaktiviert.[80] Ab 70 °C verliert das Virus endgültig seine Infektiosität.
Als behülltes Virus ist das Influenza-A-Virus empfindlich gegenüber Detergentien[81][82] und organischen Lösemitteln wie Alkoholen (z. B. Ethanol, Isopropanol).[81][82] Die metastabile Fusionsdomäne des Hämagglutinins kann durch Säuren irreversibel ausgelöst werden.[83] Darüber hinaus kann auch eine chemische oder physikalische Denaturierung zu einem Funktionsverlust der viralen Proteine führen.[84] Durch die Vielzahl an effektiven Desinfektionsmechanismen sind Influenzaviren relativ instabil im Vergleich zu anderen Viren (z. B. dem Poliovirus oder dem Hepatitis-B-Virus).
Vorkommen
[Bearbeiten | Quelltext bearbeiten]
Beim Menschen existieren die Influenzaviren und die durch sie ausgelösten Erkrankungen weltweit, allerdings kommen im Gegensatz zu den anderen Virustypen die Influenza-C- und -D-Viren nur gelegentlich vor. Während sich die Infektionen in gemäßigten Klimazonen mit zwei Hauptwellen im Oktober/November und im Februar/März manifestieren, ist die Infektionshäufigkeit in tropischen Gefilden konstant mit einem oder zwei Höhepunkten in der Regenzeit.
Meldepflicht
[Bearbeiten | Quelltext bearbeiten]In Deutschland ist nur der direkte Nachweis von Influenzaviren namentlich meldepflichtig nach § 7 des Infektionsschutzgesetzes, soweit der Nachweis auf eine akute Infektion hinweist.
In der Schweiz ist ein positiver laboranalytische Befund zu Influenzaviren (saisonale, nicht-pandemische Typen und Subtypen) meldepflichtig und zwar nach dem Epidemiengesetz (EpG) in Verbindung mit der Epidemienverordnung und Anhang 3 der Verordnung des EDI über die Meldung von Beobachtungen übertragbarer Krankheiten des Menschen. Zu einem Influenza-A-Virus des Typs HxNy (neuer Subtyp mit pandemischem Potenzial) ist sowohl ein positiver als auch ein negativer laboranalytischer Befund nach den genannten Normen meldepflichtig.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Robert Koch-Institut: Influenza Informationsportal
- WHO: Global Influenza Surveillance and Response System (GISRS) Influenza-Epidemiologie weltweit, englisch
- Influenza Research Database: FluDB Influenza-Forschungsdatenbank
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b ICTV: ICTV Taxonomy history: Akabane orthobunyavirus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35).
- ↑ ICTV Master Species List 2018b.v2. MSL #34, März 2019
- ↑ Kurt Tobler, Mathias Ackermann, Cornel Fraefel: Allgemeine Virologie (= UTB. Band 4516). Haupt Verlag, Bern 2016, ISBN 978-3-8252-4516-0, S. 50.
- ↑ Virus Metadata Resource (VMR). In: ICTV.
- ↑ a b R. A. Lamb, P. W. Choppin: The gene structure and replication of influenza virus. In: Annual Review of Biochemistry. Nr. 52, 1983, S. 467–506; doi:10.1146/annurev.bi.52.070183.002343, PMID 6351727 (englisch).
- ↑ a b c d e f J. S. Rossman, R. A. Lamb: Influenza virus assembly and budding. In: Virology. Band 411, Nummer 2, März 2011, S. 229–236, doi:10.1016/j.virol.2010.12.003, PMID 21237476, PMC 3086653 (freier Volltext) (englisch).
- ↑ Renate König et al.: Human host factors required for influenza virus replication. In: Nature. Band 463, 2010, S. 813–817; doi:10.1038/nature08699 (englisch).
- ↑ A. J. Te Velthuis, E. Fodor: Influenza virus RNA polymerase: insights into the mechanisms of viral RNA synthesis. In: Nature reviews. Microbiology. Band 14, Nr. 8, August 2016, S. 479–493; doi:10.1038/nrmicro.2016.87, PMID 27396566, PMC 4966622 (freier Volltext) (englisch).
- ↑ L. Byrd-Leotis, R. D. Cummings, D. A. Steinhauer: The Interplay between the Host Receptor and Influenza Virus Hemagglutinin and Neuraminidase. In: International journal of molecular sciences. Band 18, Nr. 7, Juli 2017, doi:10.3390/ijms18071541, PMID 28714909, PMC 5536029 (freier Volltext) (englisch).
- ↑ a b Zhi-Yong Yang, Chih-Jen Wei, Wing-Pui Kong, Lan Wu, Ling Xu, David F. Smith, Gary J. Nabel: Immunization by Avian H5 Influenza Hemagglutinin Mutants with Altered Receptor Binding Specificity. In: Science. Band 317, Nr. 5837, 10. August 2007, ISSN 0036-8075, S. 825–828, doi:10.1126/science.1135165 (englisch).
- ↑ L. Yao, C. Korteweg, W. Hsueh, J. Gu: Avian influenza receptor expression in H5N1-infected and noninfected human tissues. In: FASEB Journal. 2008, Band 22, Nr. 3, S. 733–740; PMID 17925493 (englisch).
- ↑ R. G. Webster, W. G. Laver: The origin of pandemic influenza. In: Bulletin of the World Health Organization. 1972, Band 47, Nr. 4, S. 449–452, PMID 4540994, PMC 2480853 (freier Volltext) (englisch).
- ↑ Y. Suzuki: Sialobiology of influenza: molecular mechanism of host range variation of influenza viruses. In: Biological and Pharmaceutical Bulletin. Band 28, 2005, Nr. 3, S. 399–408; doi:10.1248/bpb.28.399, PMID 15744059 (englisch).
- ↑ A. Iwai, T. Shiozaki, T. Miyazaki: Relevance of signaling molecules for apoptosis induction on influenza A virus replication. In: Biochemical and biophysical research communications. Band 441, Nummer 3, November 2013, S. 531–537; doi:10.1016/j.bbrc.2013.10.100, PMID 24177013 (englisch).
- ↑ P. Gaur, P. Ranjan, S. Sharma, J. R. Patel, J. B. Bowzard, S. K. Rahman, R. Kumari, S. Gangappa, J. M. Katz, N. J. Cox, R. B. Lal, S. Sambhara, S. K. Lal: Influenza A virus neuraminidase protein enhances cell survival through interaction with carcinoembryonic antigen-related cell adhesion molecule 6 (CEACAM6) protein. In: The Journal of biological chemistry. Band 287, Nummer 18, April 2012, S. 15109–15117, doi:10.1074/jbc.M111.328070, PMID 22396546, PMC 3340274 (freier Volltext) (englisch).
- ↑ S. E. Lindstrom, Y. Hiromoto, H. Nishimura, T. Saito, R. Nerome, K. Nerome: Comparative analysis of evolutionary mechanisms of the hemagglutinin and three internal protein genes of influenza B virus: multiple cocirculating lineages and frequent reassortment of the NP, M, and NS genes. In: Journal of virology. 1999, Band 73, Nr. 5, S. 4413–4426; PMID 10196339, PMC 104222 (freier Volltext) (englisch).
- ↑ M. R. Hilleman: Realities and enigmas of human viral influenza: pathogenesis, epidemiology and control. In: Vaccine. Band 20, 2002, Nr. 25–26, S. 3068–3087, PMID 12163258.
- ↑ K. Martin, A. Helenius: Transport of incoming Influenza Virus Nucleocapsids into the Nucleus. In: Journal of Virology. 1991, Band 56, Nr. 1, S. 232, PMID 1985199 (englisch).
- ↑ D. M. Knipe, Peter M. Howley (Hrsg.): Fields Virology. 4. Auflage, Philadelphia 2001, ISBN 0-7817-6060-7 (englisch).
- ↑ K. Haye, S. Bourmakina, T. Moran, A. García-Sastre, A. Fernandez-Sesma: The NS1 protein of a human influenza virus inhibits type I interferon production and the induction of antiviral responses in primary human dendritic and respiratory epithelial cells. In: Journal of Virology. 2009, Band 83, Nr. 13, S. 6849–6862; PMID 19403682 (englisch).
- ↑ B. G. Hale, R. E. Randall, J. Ortín, D. Jackson: The multifunctional NS1 protein of influenza A viruses. In: Journal of General Virology. 2008, Band 89, Nr. 10, S. 2359–2376; PMID 18796704 (englisch).
- ↑ N. C. Robb, M. Smith, F. T. Vreede, E. Fodor: NS2/NEP protein regulates transcription and replication of the influenza virus RNA genome. In: Journal of General Virology . 2009, Band 90, Teil 6, S. 1398–1407; PMID 19264657 (englisch).
- ↑ I. Mazur, D. Anhlan, D. Mitzner, L. Wixler, U. Schubert, S. Ludwig: The proapoptotic influenza A virus protein PB1-F2 regulates viral polymerase activity by interaction with the PB1 protein. In: Cell Microbiology. 2008, Band 10, Nr. 5, S. 1140–1152; PMID 18182088 (englisch).
- ↑ O. Haller, P. Staeheli, M. Schwemmle, G. Kochs: Mx GTPases: dynamin-like antiviral machines of innate immunity. In: Trends in microbiology. Band 23, Nr. 3, März 2015, S. 154–163; doi:10.1016/j.tim.2014.12.003, PMID 25572883 (englisch).
- ↑ M. Le Bel, J. Gosselin: Leukotriene B4 Enhances NOD2-Dependent Innate Response against Influenza Virus Infection. In: PloS one. Band 10, Nummer 10, 2015, S. e0139856; doi:10.1371/journal.pone.0139856, PMID 26444420, PMC 4596707 (freier Volltext) (englisch).
- ↑ a b W. Wu, W. Zhang, E. S. Duggan, J. L. Booth, M. H. Zou, J. P. Metcalf: RIG-I and TLR3 are both required for maximum interferon induction by influenza virus in human lung alveolar epithelial cells. In: Virology. Band 482, August 2015, S. 181–188; doi:10.1016/j.virol.2015.03.048, PMID 25880109, PMC 4461467 (freier Volltext) (englisch).
- ↑ I. K. Pang, P. S. Pillai, A. Iwasaki: Efficient influenza A virus replication in the respiratory tract requires signals from TLR7 and RIG-I. In: Proceedings of the National Academy of Sciences. Band 110, Nummer 34, August 2013, S. 13910–13915; doi:10.1073/pnas.1303275110, PMID 23918369, PMC 3752242 (freier Volltext) (englisch).
- ↑ M. G. Torcia, L. Nencioni, A. M. Clemente, L. Civitelli, I. Celestino, D. Limongi, G. Fadigati, E. Perissi, F. Cozzolino, E. Garaci, A. T. Palamara: Sex differences in the response to viral infections: TLR8 and TLR9 ligand stimulation induce higher IL10 production in males. In: PloS one. Band 7, Nummer 6, 2012, S. e39853; doi:10.1371/journal.pone.0039853, PMID 22768144, PMC 3387221 (freier Volltext) (englisch).
- ↑ R. J. Thapa, J. P. Ingram, K. B. Ragan et al.: DAI Senses Influenza A Virus Genomic RNA and Activates RIPK3-Dependent Cell Death. In: Cell host & microbe. Band 20, Nummer 5, November 2016, S. 674–681; doi:10.1016/j.chom.2016.09.014, PMID 27746097 (englisch).
- ↑ A. A. Benitez, M. Panis, J. Xue et al.: In Vivo RNAi Screening Identifies MDA5 as a Significant Contributor to the Cellular Defense against Influenza A Virus. In: Cell reports. Band 11, Nummer 11, Juni 2015, S. 1714–1726; doi:10.1016/j.celrep.2015.05.032, PMID 26074083, PMC 4586153 (freier Volltext) (englisch).
- ↑ S. Kim, M. J. Kim, D. Y. Park, H. J. Chung, C. H. Kim, J. H. Yoon, H. J. Kim: Mitochondrial reactive oxygen species modulate innate immune response to influenza A virus in human nasal epithelium. In: Antiviral research. Band 119, Juli 2015, S. 78–83; doi:10.1016/j.antiviral.2015.04.011, PMID 25930096 (englisch).
- ↑ J. D. Ong, A. Mansell, M. D. Tate: Hero turned villain: NLRP3 inflammasome-induced inflammation during influenza A virus infection. In: Journal of leukocyte biology. [elektronische Veröffentlichung vor dem Druck] Oktober 2016, doi:10.1189/jlb.4MR0616-288R, PMID 27707881 (englisch).
- ↑ A. M. Pham, F. G. Santa Maria, T. Lahiri, E. Friedman, I. J. Marié, D. E. Levy: PKR Transduces MDA5-Dependent Signals for Type I IFN Induction. In: PLoS pathogens. Band 12, Nummer 3, März 2016, S. e1005489, doi:10.1371/journal.ppat.1005489, PMID 26939124, PMC 4777437 (freier Volltext) (englisch).
- ↑ R. König, S. Stertz, Y. Zhou, A. Inoue et al.: Human host factors required for influenza virus replication. In: Nature. Band 463, Nummer 7282, Februar 2010, S. 813–817; doi:10.1038/nature08699, PMID 20027183, PMC 2862546 (freier Volltext) (englisch).
- ↑ Franz X. Heinz: Dem Geheimnis der Influenza Pandemien auf der Spur. Abgerufen am 9. Januar 2013.
- ↑ Maria Jose Alonso: Nanostructured Biomaterials for Overcoming Biological Barriers. Royal Society of Chemistry, 2012, ISBN 978-1-84973-529-2, S. 161 (englisch).
- ↑ I. Banerjee, Y. Miyake, S. P. Nobs et al.: Influenza A virus uses the aggresome processing machinery for host cell entry. In: Science. Band 346, Nummer 6208, Oktober 2014, S. 473–477, ISSN 1095-9203. doi:10.1126/science.1257037. PMID 25342804 (englisch).
- ↑ A. Stevaert, L. Naesens: The Influenza Virus Polymerase Complex: An Update on Its Structure, Functions, and Significance for Antiviral Drug Design. In: Medicinal research reviews. Band 36, Nr. 6, 11 2016, S. 1127–1173; doi:10.1002/med.21401, PMID 27569399, PMC 5108440 (freier Volltext) (englisch).
- ↑ a b c A. Pflug, M. Lukarska, P. Resa-Infante, S. Reich, S. Cusack: Structural insights into RNA synthesis by the influenza virus transcription-replication machine. In: Virus research. Band 234, April 2017, S. 103–117; doi:10.1016/j.virusres.2017.01.013, PMID 28115197 (englisch).
- ↑ E. Fodor: The RNA polymerase of influenza a virus: mechanisms of viral transcription and replication. In: Acta virologica. Band 57, Nr. 2, 2013, S. 113–122; PMID 23600869 (englisch).
- ↑ J. Reguera, P. Gerlach, S. Cusack: Towards a structural understanding of RNA synthesis by negative strand RNA viral polymerases. In: Current opinion in structural biology. Band 36, Februar 2016, S. 75–84; doi:10.1016/j.sbi.2016.01.002, PMID 26826467 (englisch).
- ↑ E. W. Brydon, S. J. Morris, C. Sweet: Role of apoptosis and cytokines in influenza virus morbidity. (PDF; 505 kB) In: FEMS microbiology reviews. Band 29, Nr. 4, September 2005, S. 837–850; doi:10.1016/j.femsre.2004.12.003, PMID 16102605 (englisch).
- ↑ S. J. Stray, G. M. Air: Apoptosis by influenza viruses correlates with efficiency of viral mRNA synthesis. In: Virus research. Band 77, Nr. 1, September 2001, ISSN 0168-1702, S. 3–17, PMID 11451482 (englisch).
- ↑ L. Möhler, D. Flockerzi, H. Sann, U. Reichl: Mathematical model of influenza A virus production in large-scale microcarrier culture. In: Biotechnology and bioengineering. Band 90, Nr. 1, April 2005, ISSN 0006-3592, S. 46–58, doi:10.1002/bit.20363, PMID 15736163 (englisch).
- ↑ H. J. Cairns, M. Edney, S. Fazekas de St Groth: Quantitative aspects of influenza virus multiplication. In: Journal of Immunology (Baltimore, Md.: 1950). Band 69, Nummer 2, August 1952, ISSN 0022-1767, S. 155–181, PMID 14946411 (englisch).
- ↑ WHO: A revision of the system of nomenclature for influenza viruses: a WHO memorandum. In: Bulletin of the World Health Organization. Band 58, Nr 4, 1980, S. 585–591, PMID 6969132, PMC 2395936 (freier Volltext) (englisch).
- ↑ a b WHO: Influenza virus infections in humans (February 2014) (PDF; 298 kB) Abgerufen am 2. März 2018.
- ↑ WHO: Standardization of terminology for the influenza virus variants infecting humans: Update. Joint announcement of FAO, OIE and WHO. Vom 30. Januar 2014. Abgerufen am 2. März 2018.
- ↑ M. J. Pantin-Jackwood, D. E. Swayne: Pathogenesis and pathobiology of avian influenza virus infection in birds. In: Scientific and Technical Review. Band 28, Nr. 1, 2009, S. 113–136; PMID 19618622 (englisch).
- ↑ T. W. Vahlenkamp, T. C. Harder: Influenza virus infections in mammals. In: erliner und Münchener Tierärztliche Wochenschrift. Band 119, 2006, Nr. 3–4, S. 123–131; PMID 16573202 (englisch).
- ↑ H. M. Yassine, C. W. Lee, R. Gourapura, Y. M. Saif: Interspecies and intraspecies transmission of influenza A viruses: viral, host and environmental factors. In: Animal Health Research Reviews. Band 11, 2010, Nr. 1, S. 53–72; PMID 20591213 (englisch).
- ↑ Marcel G. E. Rommel, Lisa Walz, Foteini Fotopoulou, Saskia Kohlscheen, Franziska Schenk et al.: Influenza A virus infection instructs hematopoiesis to megakaryocyte-lineage output.In: Cell reports. 4. Oktober 2022, Band 4, Nr. 41(1), S. 111447; doi:10.1016/j.celrep.2022.111447, PMID 36198277 (englisch).
- ↑ NCBI: Influenza A virus (no rank).
- ↑ Informationen zur Vogelgrippe ( vom 30. März 2009 im Internet Archive).
- ↑ Richard E. Shope: Swine Influenza: III. Filtration and Ion Experiments and Etiology. In: Journal of Experimental Medicine. Band 54, 1931, S. 373–385.
- ↑ Suburban Emergency Management Project ( vom 4. März 2010 im Internet Archive), Biot Report 162 vom 9. Januar 2005: What Is Swine Flu?
- ↑ H3N2 in der Influenza Research Database.
- ↑ World Organisation for Animal Health ( vom 2. Februar 2014 im Internet Archive) vom 4. April 2013: Low pathogenic avian influenza (poultry), China (People’s Rep. of). Information received on 04/04/2013 from Dr Zhang Zhongqui, Director General, China Animal Disease Control Centre, Veterinary Bureau, Ministry of Agriculture, Beijing, China (People’s Rep. of).
- ↑ Xian Qi, Yan-Hua Qian, Chang-Jun Bao et al.: Probable person to person transmission of novel avian influenza A (H7N9) virus in Eastern China, 2013: epidemiological investigation. In: British Medical Journal. (BMJ) 6. August 2013, Band 347, S. 4752, doi:10.1136/bmj.f4752 (englisch).
- ↑ Das RKI zu humanen Erkrankungsfällen mit aviärer Influenza A(H7N9). Auf: rki.de, Stand: 24. Mai 2018; abgerufen am 19. April 2019.
- ↑ R. J. Webby, R. G. Webster, J. A. Richt: Influenza viruses in animal wildlife populations. In: Current Topics in Microbiology and Immunology. Nr. 315, 2007, S. 67–83, PMID 17848061.
- ↑ A. D. Osterhaus, G. F. Rimmelzwaan, B. E. Martina, T. M. Bestebroer, R.A. Fouchier: Influenza B virus in seals. In: Science. 288. Jahrgang, Nr. 5468, 2000, S. 1051–1053, doi:10.1126/science.288.5468.1051, PMID 10807575 (englisch)..
- ↑ R. Bodewes, D. Morick, G. de Mutsert, N. Osinga, T. Bestebroer, S. van der Vliet, S. L. Smits, T. Kuiken, G. F. Rimmelzwaan, R. A. Fouchier, A. D. Osterhaus: Recurring influenza B virus infections in seals. In: Emerging Infectious Diseases. 19. Jahrgang, Nr. 3, 2013, S. 511–512, doi:10.3201/eid1903.120965, PMID 23750359 (englisch)..
- ↑ NCBI: Influenza B virus (spezies)
- ↑ NCBI: Influenza B virus, Victoria strains.
- ↑ NCBI: Influenza B virus, Yamagata strains.
- ↑ NCBI: Influenza B virus, Yamaguchi strains.
- ↑ NCBI: Influenza B virus, Yokohama strains.
- ↑ NCBI: Influenza B virus, Yunnan strains.
- ↑ NCBI: Influenza B virus, Zhuhai strains.
- ↑ NCBI: Influenza C virus (spezies).
- ↑ B. M. Hause, M. Ducatez, E. A. Collin, Z. Ran et al.: Isolation of a novel swine influenza virus from Oklahoma in 2011 which is distantly related to human influenza C viruses. In: PLOS Pathogens (PLoS Pathog). 9. Jahrgang, Nr. 2, 2013, doi:10.1371/journal.ppat.1003176 (englisch)..
- ↑ Z. Sheng, Z. Ran, D. Wang et al.: Genomic and evolutionary characterization of a novel influenza-C-like virus from swine. In: Archives of Virology. 159. Jahrgang, Nr. 2, 2014, S. 249–255, doi:10.1007/s00705-013-1815-3 (englisch)..
- ↑ Deltainfluenzavirus, auf: ViralZone.
- ↑ E. A. Collin, Z. Sheng, Y. Lang, W. Ma, B. M. Hause, F. Li: Cocirculation of two distinct genetic and antigenic lineages of proposed influenza D virus in cattle. In: Journal of Virology. 89. Jahrgang, Nr. 2, Januar 2015, S. 1036–1042, doi:10.1128/JVI.02718-14 (englisch)..
- ↑ NCBI: Influenza D virus (spezies).
- ↑ M. S. Miller, T. J. Gardner, F. Krammer, L. C. Aguado, D. Tortorella, C. F. Basler, P. Palese: Neutralizing Antibodies Against Previously Encountered Influenza Virus Strains Increase over Time: A Longitudinal Analysis. In: Science Translational Medicine. Nr. 5, 2013, S. 198ra107–198ra107, doi:10.1126/scitranslmed.3006637 (englisch).
- ↑ J. D. Parvin, A. Moscona, W. T. Pan, J. M. Leider, P. Palese: Measurement of the mutation rates of animal viruses: influenza A virus and poliovirus type 1. In: Journal of virology. Band 59, Nr. 2, August 1986, ISSN 0022-538X, S. 377–383, PMID 3016304, PMC 253087 (freier Volltext) (englisch).
- ↑ Ivan V. Polozov, Ludmila Bezrukov, Klaus Gawrisch, Joshua Zimmerberg: Progressive ordering with decreasing temperature of the phospholipids of influenza virus. In: Nature Chemical Biology. Band 4, 2008, S. 248–255, PMID 18311130 (englisch).
- ↑ Akademie für öffentliches Gesundheitswesen Düsseldorf (Hrsg.): Blickpunkt öffentliche Gesundheit. Nr. 04/2001.
- ↑ a b M. L. Grayson, S. Melvani, J. Druce, I. G. Barr, S. A. Ballard, P. D. Johnson, T. Mastorakos, C. Birch: Efficacy of soap and water and alcohol-based hand-rub preparations against live H1N1 influenza virus on the hands of human volunteers. In: Clinical Infectious Diseases. 2009, Band 48, Nr. 3, S. 285–291; PMID 19115974 (englisch).
- ↑ a b E. K. Jeong, J. E. Bae, I. S. Kim: Inactivation of influenza A virus H1N1 by disinfection process. In: American Journal of Infection Control. 2010, Band 38, Nr. 5, S. 354–360; PMID 20430477 (englisch).
- ↑ M. Nishide, K. Tsujimoto, M. Uozaki, K. Ikeda, H. Yamasaki, A. H. Koyama, T. Arakawa: Effects of electrolytes on virus inactivation by acidic solutions. In: International Journal of Molecular Medicine. 2011, Band 27, Nr. 6, S. 803–809; doi:10.3892/ijmm.2011.668, PMID 21468540 (englisch).
- ↑ M. Abe, K. Kaneko, A. Ueda, H. Otsuka, K. Shiosaki, C. Nozaki, S. Goto: Effects of several virucidal agents on inactivation of influenza, Newcastle disease, and avian infectious bronchitis viruses in the allantoic fluid of chicken eggs. In: Japanese Journal of Infectious Diseases. 2007, Band 60, Nr. 6, S. 342–346; PMID 18032831 (englisch).




